Bulk RNA-seq与scRNA-seq的优势和局限性
Bulk RNA-seq
- 20世纪末即芯片技术后的又一个突破性技术,之后被广泛应用,逐渐替代芯片
- 评估测量一个大群体内所有细胞的平均表达量水平
- 适用于比较转录组学,如不同物种相同组织的不同样本间的转录表达差异比较;或者用于评估疾病等表达量特征
- 不适用于研究系统的异质性,如早期胚胎发育、复杂组织(脑)
- 不能提供基因表达量的随机特征
scRNA-seq
- 2009年出现 (Tang et al. 2009)
- 伴随着新的protocols和测序成本的降低,2014年左右被广泛推广
- 适用于检测一群细胞的表达量分布水平;细胞特异性变化等(如鉴定细胞类型、细胞应激的异质性,基因表达的随机性、推测细胞间的基因调控网络
- 数据集从102~ 10 8细胞,且逐年增长
- 目前的使用的protocols:SMART-seq2 (Picelli et al. 2013), CELL-seq (Hashimshony et al. 2012) and Drop-seq (Macosko et al. 2015)
- 商业平台:Fluidigm C1, Wafergen ICELL8 and the 10X Genomics Chromium
- Bulk RNA-seq的一些方法也适合于scRNA-seq,大多情况需要新的方法
单细胞RNA测序流程
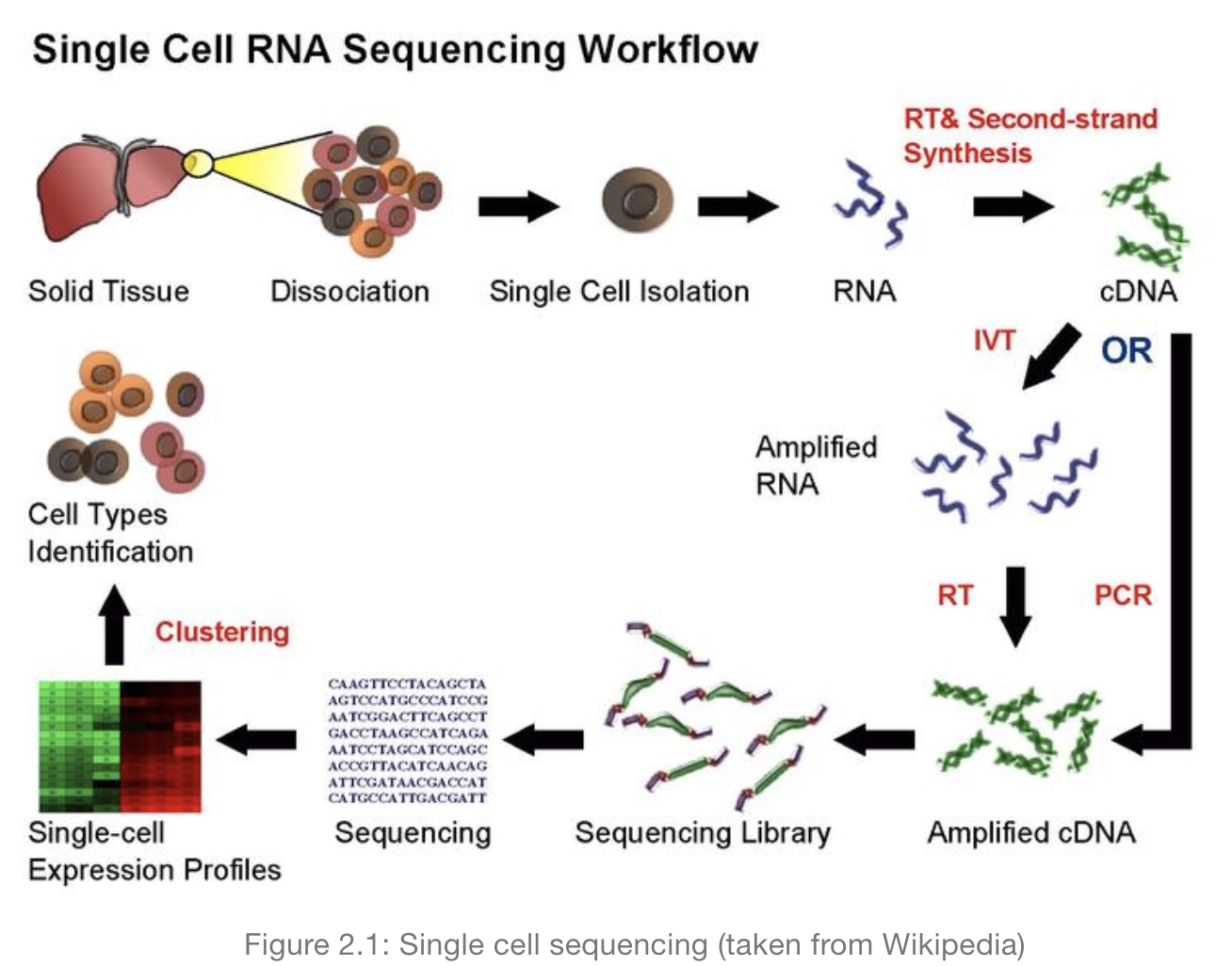
分析方法
scRNA-seq上游分析方法与bulk 相同,对测序数据进行质控、比对、比对结果的质控(黄色部分),接着是细胞质控、标准化,这一部分与bulk有相同的也有不同的地方,下游分析就是解释实际生物学问题,包括差异分析、聚类、网络分析等,所用的方法一般都是scRNA特有的算法。
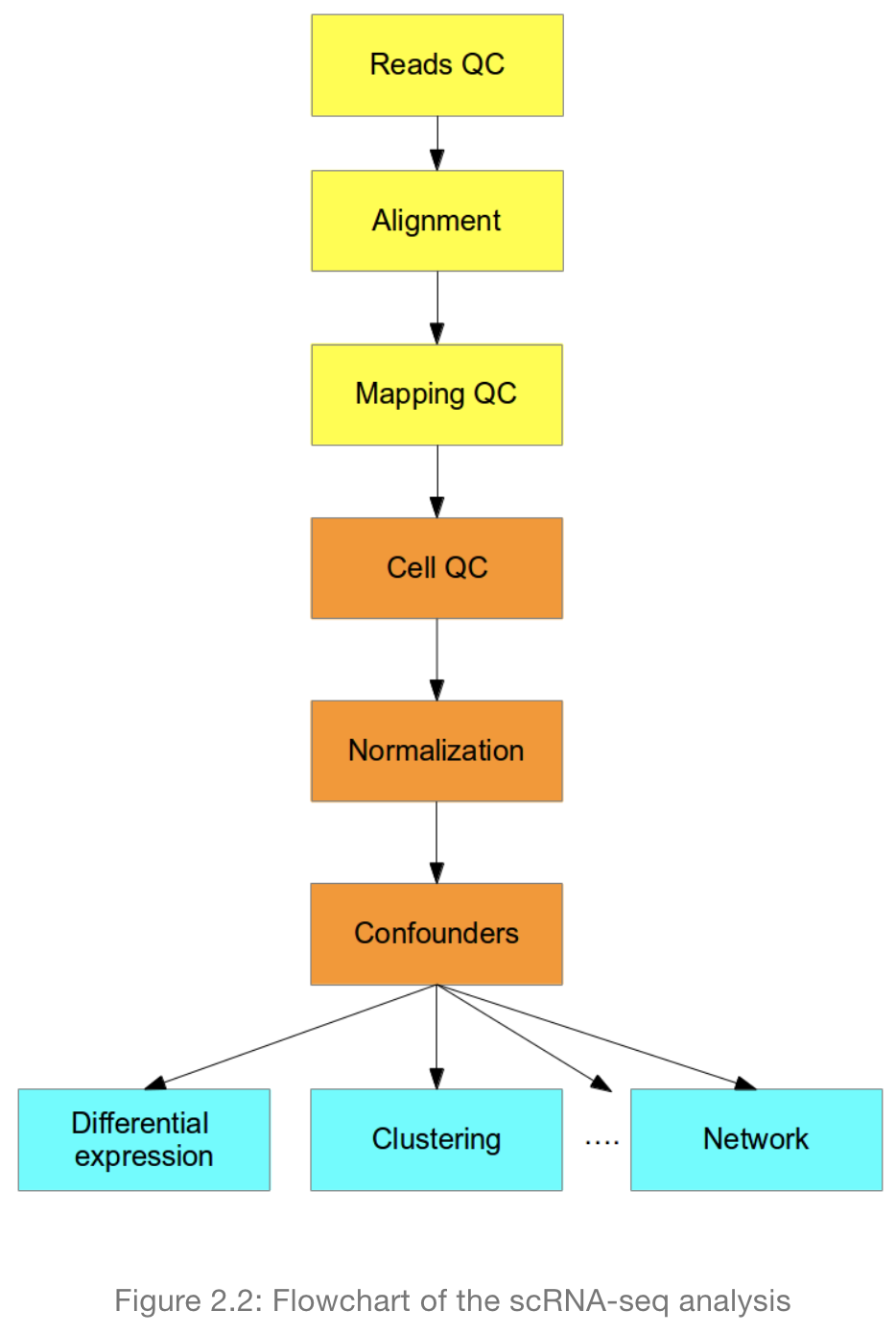
scRNA-seq 分析的综述:(Stegle, Teichmann, and Marioni 2015)
- Falco :云端处理scRNA-seq分析.
- SCONE (Single-Cell Overview of Normalized Expression), scRNA-seq质控和标准化的R包
- Seurat :scRNA-seq分析的常用的四大R包之一,包括对数据的质控、分析等
- ASAP (Automated Single-cell Analysis Pipeline) :scRNA-seq交互分析的网络平台
挑战
bulk RNA-seq与scRNA-seq最主要的不同是scRNA-seq的每个测序文库代表一个细胞而不是一群细胞。文库的不同来源于:
- 扩增 (达到1 million倍)
- Gene ‘dropouts’:指一个细胞在一个细胞中是中等表达水平,但是在另一个细胞中没有被检测到
上面的差异主要来自于RNA的起始量很低,因此提高转录本的捕获率、降低扩增偏差是一个热门的研究方向。
实验方法
发展新的scRNA-seq protocol也是一个热门领域,目前已有的方法包括:
- CEL-seq (Hashimshony et al. 2012)
- CEL-seq2 (Hashimshony et al. 2016)
- Drop-seq (Macosko et al. 2015)
- InDrop-seq (Klein et al. 2015)
- MARS-seq (Jaitin et al. 2014)
- SCRB-seq (Soumillon et al. 2014)
- Seq-well (Gierahn et al. 2017)
- Smart-seq (Picelli et al. 2014)
- Smart-seq2 (Picelli et al. 2014)
- SMARTer
- STRT-seq (Islam et al. 2013)
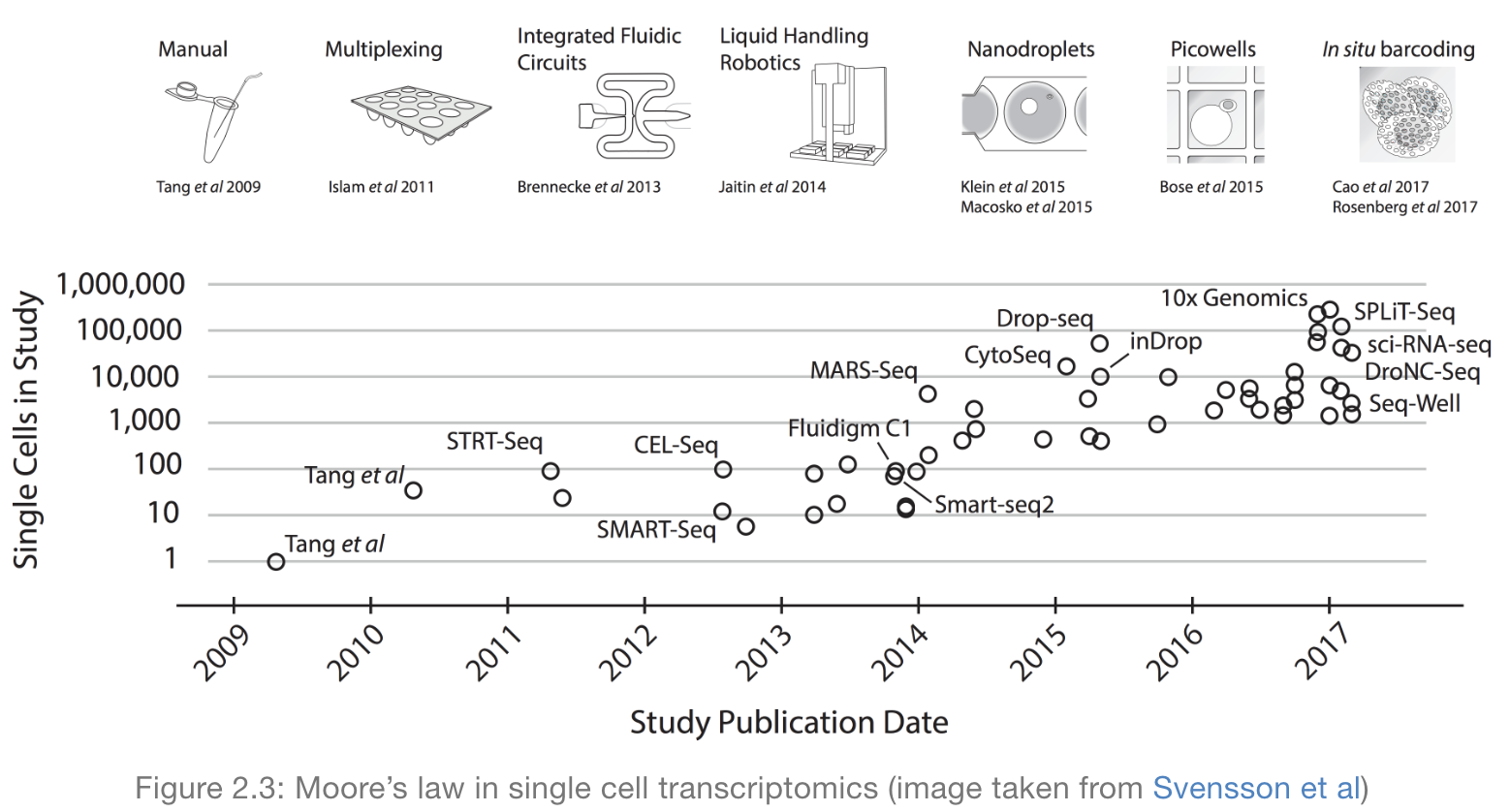
不同的方法实现方式不同,其中定量和捕获是最重要的两个方面
定量有两种类型:
- 全长(full length):
- 致力于实现每个转录本一致的read coverage
- 理论上的一致的coverage很难实现,经常会存在偏差
- 基于标签(tag-based):
- 只捕获RNA的5‘ 端或3’端,
- 可以和UMI(unique molecular identifiers )结合提高定量准确性
- 由于只捕获单端RNA可能会降低比对率
- 很难鉴定isoforms
捕获:
捕获策略决定了测序通量,以及得到细胞的方式和其他信息。常用的捕获策略有:
microwell-based
使用吸液管或激光捕获等方法分离细胞,并将其放置在微流体管中。该方法的优缺点是:
优点是:可以与FACS分选结合使用,从而可以根据细胞表面marker筛选感兴趣的特定类型的细胞,同时在分选时可以拍摄细胞。图像可以用于识别受损细胞或dublets。
缺点是:通量低、耗时费工

microfluidic-based
如Fluidigm’s C1,提供了一个更为集成的系统,用于捕捉细胞和进行建库准备所需的反应。
优点是:通量比microwell-based 高
缺点是:捕获效率低,只有10%的细胞可以被捕获,因此不适用于细胞类型罕见或者输入细胞量少的研究;另外芯片价格相对贵

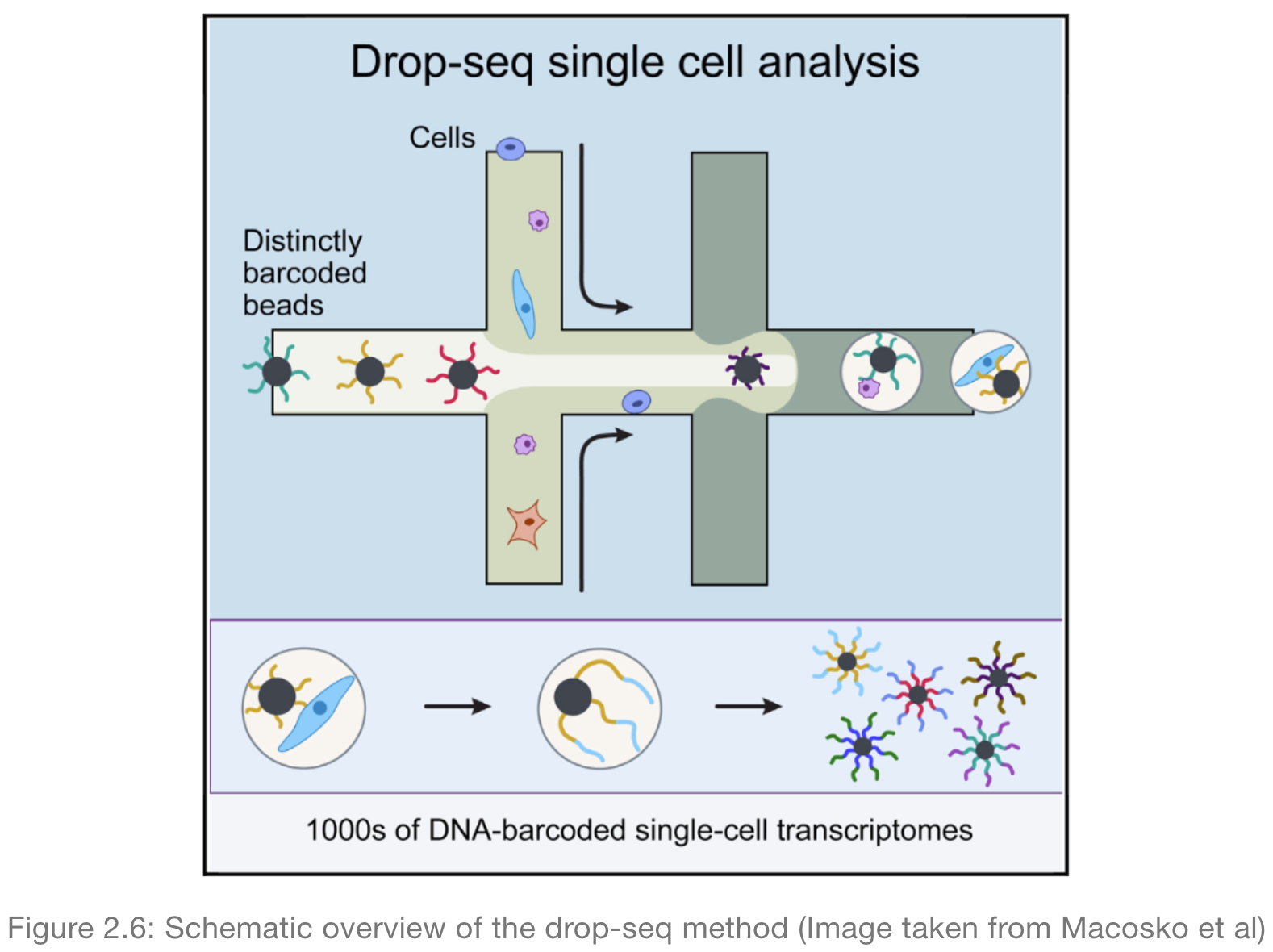
droplet- based
将每一个细胞封装在一个毫微升的液滴中,并加上一个珠子(bead), bead中含有建库所需的酶和唯一的条形码(barcode),条形码可以将reads来自于哪个细胞关联。因此,所有的液滴都可以汇集起来,排序在一起,然后根据条形码将reads分配给原始细胞。
优点是:通量最高
缺点是:测序成本、低覆盖率 (Ziegenhain et al. 2017)
如何选择合适的平台
如何选择合适的protocols,只有根据研究的生物学问题。如果感兴趣一个组织的组成,那么droplet-based的方法可以得到大量的细胞,或许是最适合的;如果研究的是罕见细胞,并且知道细胞marker,那么结合FACS富集,测少量的细胞也可以实现目的。
全长转录本适合于对不同亚型感兴趣的研究,tagged的protocols可以和UMI结合,适用于基因水平的定量。
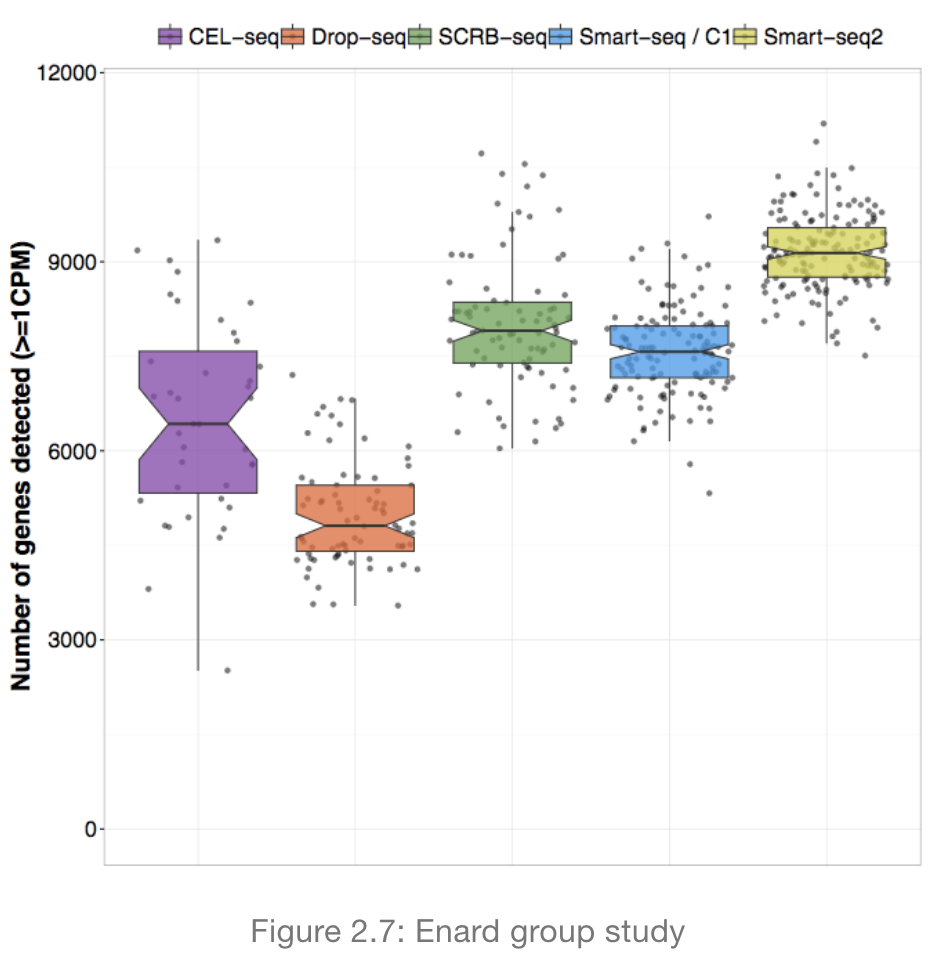
内容来自Hemberg lab’s 单细胞课程学习笔记 :
Introduction to single-cell RNA-seq:
https://scrnaseq-course.cog.sanger.ac.uk/website/introduction-to-single-cell-rna-seq.html