一句话评价
scATAC-seq分析方法评估

文章信息
题目:Assessment of computational methods for the analysis of single-cell ATAC-seq data
杂志:bioRxiv
时间:Aug.18,2019
链接: DOI: http://dx.doi.org/10.1101/739011.
文章介绍:
scATAC-seq技术目前广泛应用于不同领域转录调控的研究,伴随着很多计算方法相继被开发,用于分析、解释scATAC-seq数据背后的生物学意义。这篇文章对scATAC-seq分析的10个工具在13个数据集中(包括3个真实数据集和10个合成数据集)进行了测评。
10个工具分别是:
- BROCKMAN
- de Boer, C.G. and A. Regev, BROCKMAN: deciphering variance in epigenomic regulators by k-mer factorization. BMC Bioinformatics, 2018. 19(1): p. 253.
- chromVAR
- Schep, A.N., et al., chromVAR: inferring transcription-factor-associated accessibility from single-cell epigenomic data. Nat Methods, 2017. 14(10): p. 975-978.
- Cicero
- Pliner, H.A., et al., Cicero Predicts cis-Regulatory DNA Interactions from Single-Cell Chromatin Accessibility Data. Mol Cell, 2018. 71(5): p. 858-871 e8.
- cisTopic
- Bravo González-Blas, C., et al., cisTopic: cis-regulatory topic modeling on single-cell ATAC-seq data. Nature Methods, 2019.
- Cusanovich2018
- Cusanovich, D.A., et al., A Single-Cell Atlas of In Vivo Mammalian Chromatin Accessibility. Cell, 2018. 174(5): p. 1309-1324 e18.
- Cusanovich, D.A., et al., The cis-regulatory dynamics of embryonic development at single-cell resolution. Nature, 2018. 555(7697): p. 538-542.
- Cusanovich, D.A., et al., Multiplex single cell profiling of chromatin accessibility by combinatorial cellular indexing. Science, 2015. 348(6237): p. 910-4.
- Gene Scoring
- Lareau, C.A., et al., Droplet-based combinatorial indexing for massive scale single-cell epigenomics. bioRxiv, 2019: p. 612713.
- scABC
- Zamanighomi, M., et al., Unsupervised clustering and epigenetic classification of single cells. Nat Commun, 2018. 9(1): p. 2410.
- Scasat
- Baker, S.M., et al., Classifying cells with Scasat, a single-cell ATAC-seq analysis tool.Nucleic Acids Res, 2019. 47(2): p. e10.
- SCRAT
- Ji, Z., W. Zhou, and H. Ji, Single-cell regulome data analysis by SCRAT. Bioinformatics, 2017. 33(18): p. 2930-2932.
- SnapATAC
- Fang, R., et al., Fast and Accurate Clustering of Single Cell Epigenomes Reveals Cis- Regulatory Elements in Rare Cell Types. BioRxiv, 2019.
测评方法:
利用收集到的3个公共数据集和10个合成模仿的不同覆盖度和噪音水平的数据集,从bam格式的比对文件开始,1)根据10种scATAC-seq工具原文提供的方法构建下游分析所需的特征矩阵;2)然后用3种常见的聚类方法K-means、Louvain、Hierarchical clustering和UMAP找出潜在的亚群,并可视化每种方法的细胞间相似性;3)对聚类结果进行评估,有FACS分选标记和有金标准的组织采用ARI(adjusted random)、AMI(adjusted mutual information)和H(homogeneity)的方法评估聚类结果,当只知道已知的marker 基因,采用RAGI (Gini-index-based metric called Residual Average Gini Index)的方法评估。4)最后对基于第3步得到的值,对每种方法的聚类效果进行打分排序。流程如下图所示:
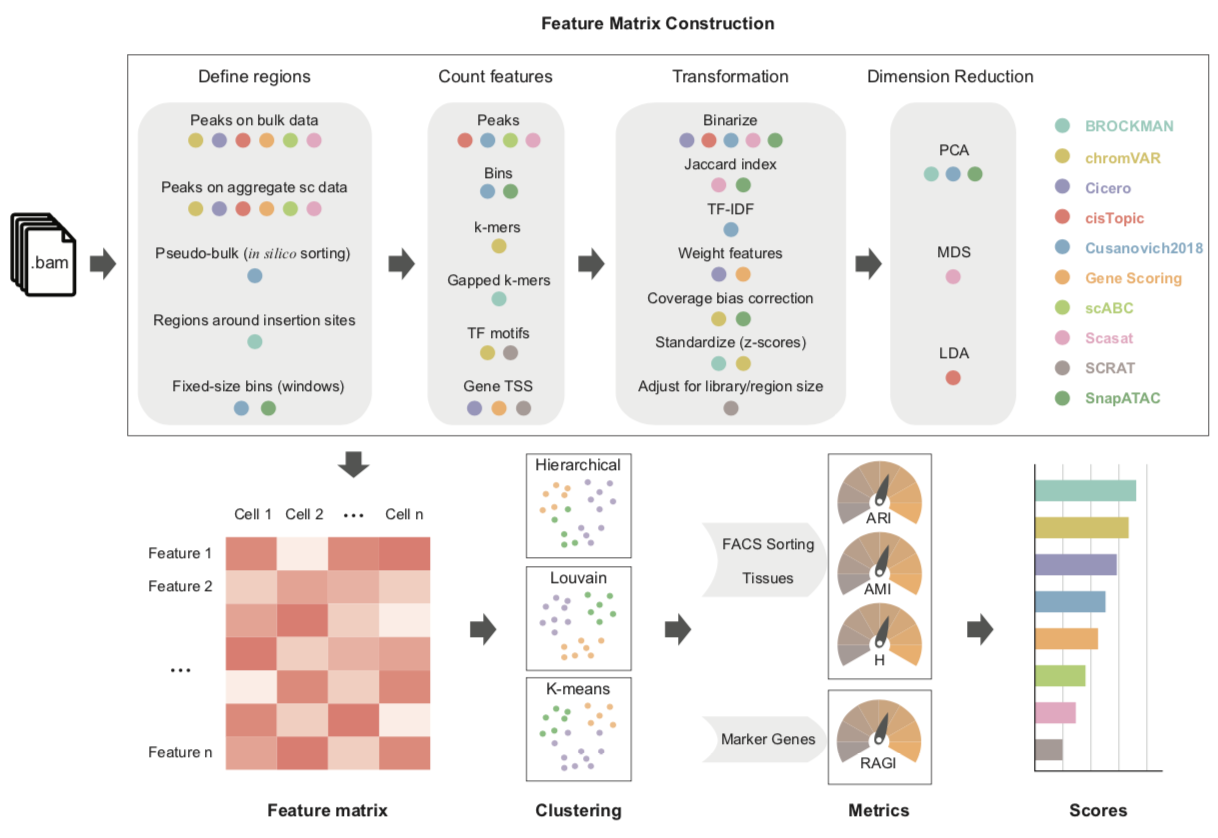
测评结果:
他们的测评结果显示SnapATAC、cisTopic和Cusanovich2018是性能最好的对scATAC-seq数据聚类分析方法。另外基于peak-level(cisTopic, Cusanovich2018, Scasat)和bin-level(SnapATAC)保存信息的方法优于motif/k-mer level (chromVAR,BROCKMAN, SCRAT)或gene-body (Cicero, Gene Scoring) 。实现降维步骤的方法(BROCKMAN、cisTopic、Cusanovich2018、Scasat、SnapATAC)通常比没有这个重要步骤的其他方法显示出优势。此外,SnapATAC是最具伸缩性的方法,它是唯一能够处理超过80000个细胞的方法。CusanovicH2018是平衡分析性能和运行时间的最佳方法。下图展示了每种方法的聚类效果评估等级和时间需求。
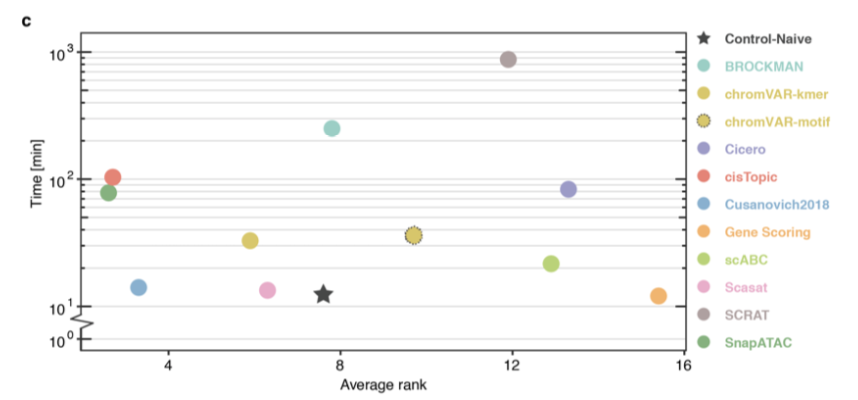
参考价值:
概述了scATAC-seq常见的分析流程
如,建库方法常用的两种策略:Droplet-based(10X genomics, BioRad), split-pool(sciATA-seq)。下游常见分析:可视化、聚类、轨迹分析、差异可及性分析、顺式调控网络。
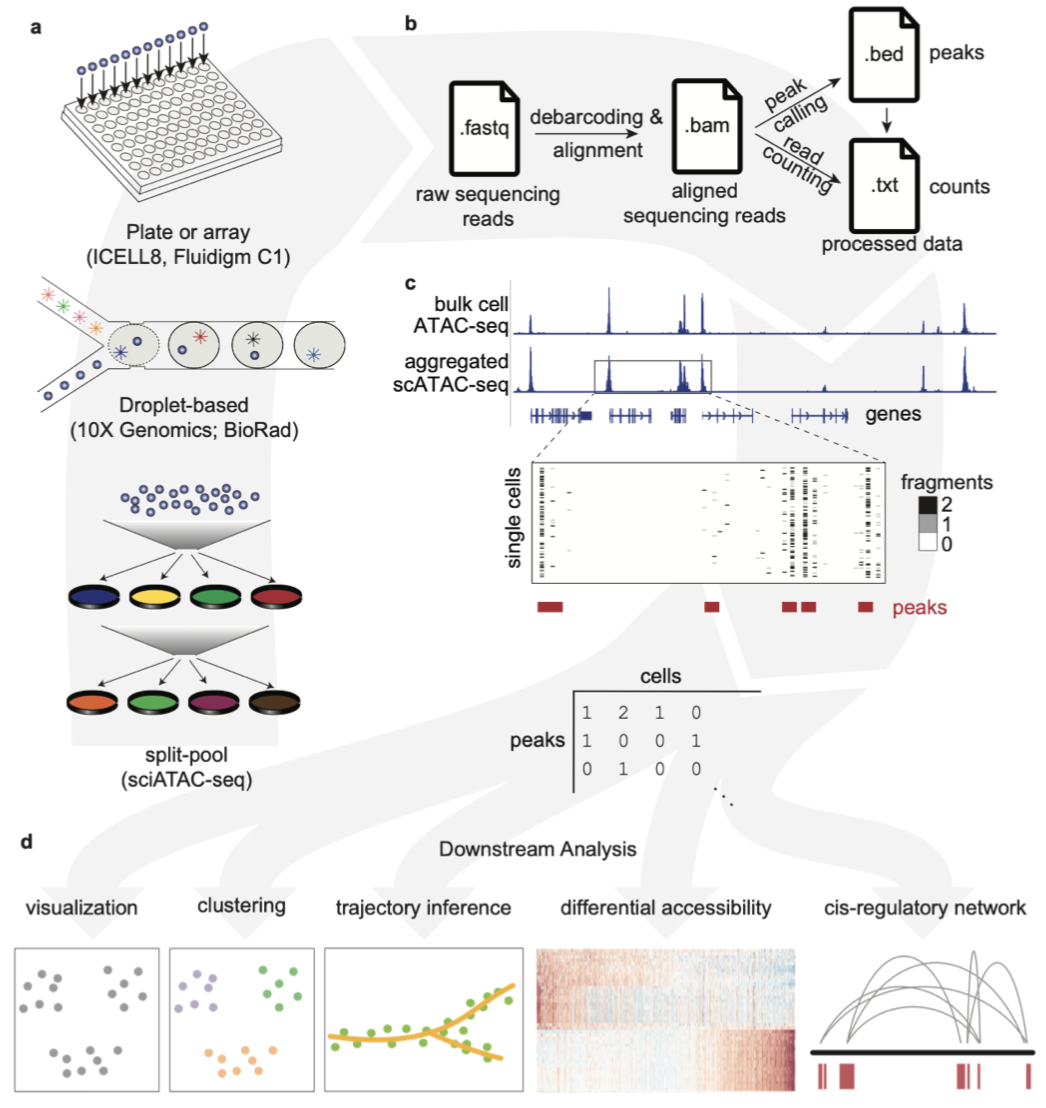
比较了10种scATAC-seq分析方法,为选择合适的方法提供了重要的参考依据
github提供了作者的100多个测试流程Jupyter笔记本,不仅可以复现作者的测评结果,还可以学习每种scATAC-seq的分析方法
github: https://github.com/pinellolab/scATAC-benchmarking/
不足:
本文的测评主要基于不同方法对scATAC-seq数据的聚类效果,但是,实际项目中可能不仅关注聚类分群,还关注谱系发育轨迹、转录因子结合位点、重要的marker基因以及他们的调控方式。
作者在讨论中也对测评的局限性做了讨论,如,chromVAR更适合于轨迹预测,chromVAR,Cicero, Gene Scoring的设计主要是为了确定关键marker基因以及他们的调控方式等。所以具体选择哪种方法,还要结合实际的研究目的决定。
碎碎念
找时间实践下github上给出的几种scATAC-seq方法
每日文献摘要:第15篇 2019年11月02日 周六