1. 基因组学和医学研究中的数据主权
题目:Data sovereignty in genomics and medical research
杂志:Nature Machine Intelligence
IF: 15
时间:17 November 2022
链接:https://www.nature.com/articles/s42256-022-00578-1
摘要
人工智能有望为医疗保健和研究带来许多好处,但是不同群体,不同国家越来越重视医学数据的隐私性和安全性,数据的共享面临着诸多挑战。为了改变这种情况,需要满足数据提供者能够控制他们自己的数据以及如何分享这些数据。目前的解决的方案有联邦学习(Federated learning),以及2021年新提出的蜂群学习(swarm learning)。
2. 野外多语言的视觉语音识别
题目:Visual speech recognition for multiple languages in the wild
杂志:Nature Machine Intelligence
IF: 15
时间:24 October 2022
链接:https://www.nature.com/articles/s42256-022-00550-z
摘要
视觉语音识别(Visual speech recognition, VSR)的目的是根据嘴唇的运动来识别语音的内容,而不依赖音频流。深度学习的进展和大型视听数据集的可用性导致了比以往任何时候都更准确和稳健的VSR模型的发展。然而,这些进步通常是由于更大的训练集,而不是模型设计。在这里,我们证明设计更好的模型与使用更大的训练集同样重要。我们建议在VSR模型中增加基于预测的辅助任务,并强调超参数优化和适当的数据增强的重要性。我们表明,这样的模型适用于不同的语言,并在很大程度上超过了以前所有在公开的数据集上训练的方法。它甚至超过了在非公开数据集上训练的模型,这些数据集的数据量最多可以达到21倍。此外,我们还表明,使用额外的训练数据,甚至是其他语言的数据或自动生成的转录,都能带来进一步的改进。
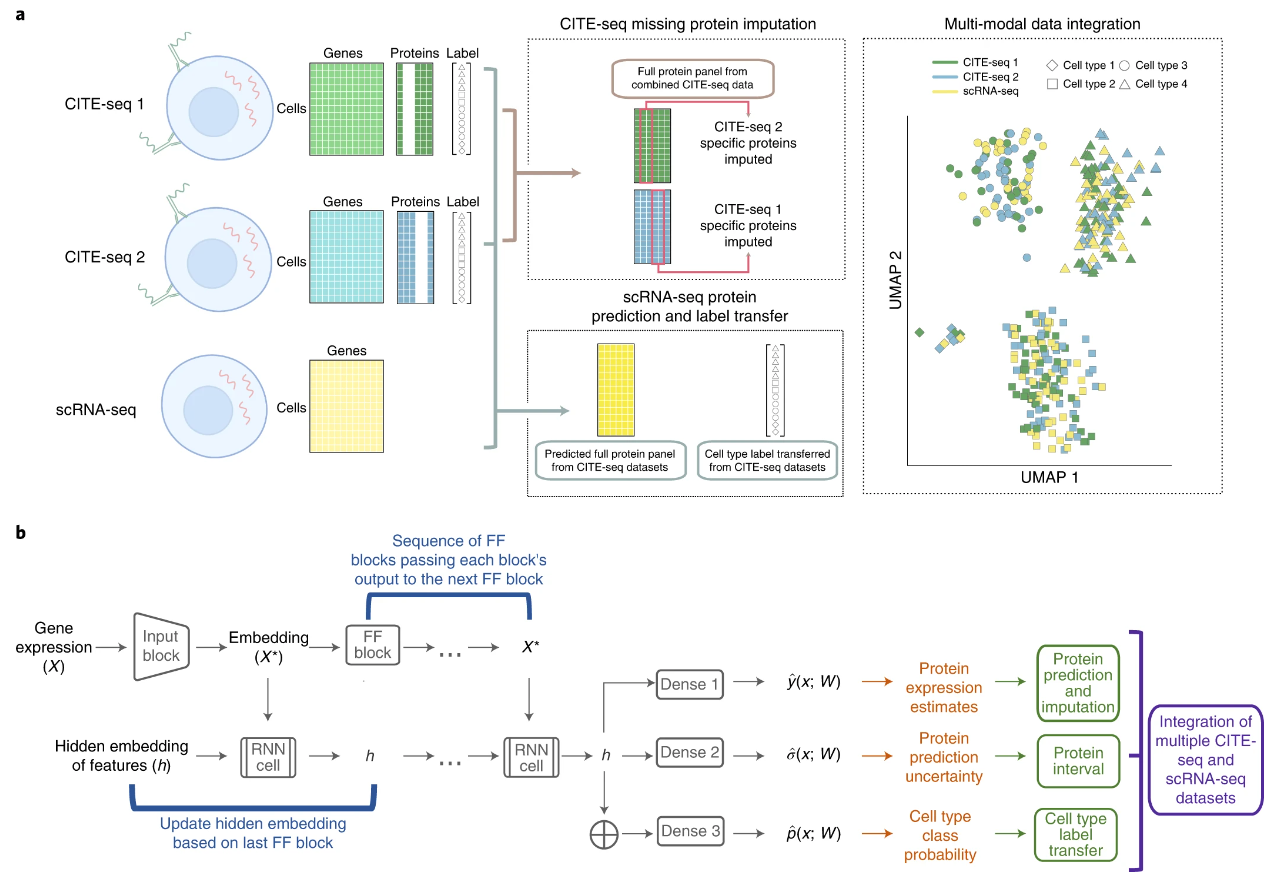
3. 用于CITE-seq和单细胞RNA-seq数据整合与细胞表面蛋白预测和归纳的多用深度学习方法
题目:A multi-use deep learning method for CITE-seq and single-cell RNA-seq data integration with cell surface protein prediction and imputation
杂志:Nature Machine Intelligence
IF: 15
时间:27 October 2022
链接:https://www.nature.com/articles/s42256-022-00545-w
摘要
CITE-seq是一种单细胞多组学技术,可同时测量单细胞中的RNA和蛋白质表达,已被广泛应用于生物医学研究,特别是免疫相关疾病和其他疾病,如流感和COVID-19。尽管CITE-seq激增,但产生这种数据的成本仍然很高。虽然数据整合可以增加信息含量,但这也带来了计算上的挑战。首先,结合多个数据集容易产生批量效应,需要加以解决。第二,很难结合多个CITE-seq数据集,因为不同数据集的蛋白质面板可能只是部分重叠。整合多个CITE-seq和单细胞RNA测序(scRNA-seq)数据集很重要,因为这样可以利用尽可能多的数据来发现细胞群的异质性。为了克服这些挑战,我们提出了sciPENN,这是一种多用途的深度学习方法,支持CITE-seq和scRNA-seq数据整合、scRNA-seq的蛋白质表达预测、CITE-seq的蛋白质表达推算、预测和推算的不确定性量化,以及从CITE-seq到scRNA-seq的细胞类型标签转移。跨越多个数据集的综合评估表明,sciPENN优于目前其他最先进的方法。
4. 通过自适应图神经网络预测未见过的抗体的中和能力
题目:Predicting unseen antibodies’ neutralizability via adaptive graph neural networks
杂志:Nature Machine Intelligence
IF: 15
时间:07 November 2022
链接:https://www.nature.com/articles/s42256-022-00553-w
摘要
大多数天然和合成抗体是 “看不见的”。也就是说,要证明它们与任何抗原的中和作用需要费力和昂贵的湿式实验室实验。现有的从已知的抗体-抗原相互作用中学习抗体表征的方法,由于缺乏相互作用的实例,不适合于未见过的抗体。本文提出的DeepAAI方法通过构建抗体和抗原之间的两个适应性关系图,并在未见过的和见过的抗体表征之间应用拉普拉斯平滑法来学习未见过的抗体表征。DeepAAI不是使用静态的蛋白质描述符,而是 “动态地 “学习表征和关系图,并针对中和预测和50%抑制浓度估计的下游任务进行优化。DeepAAI的性能在人类免疫缺陷病毒、严重急性呼吸道综合征冠状病毒2、流感和登革热上得到了证明。此外,关系图具有丰富的可解释性。抗体关系图意味着抗体中和反应的相似性,而抗原关系图表明病毒不同变体之间的关系。因此,我们建议对这些病毒的新变体采用可能的广谱抗体。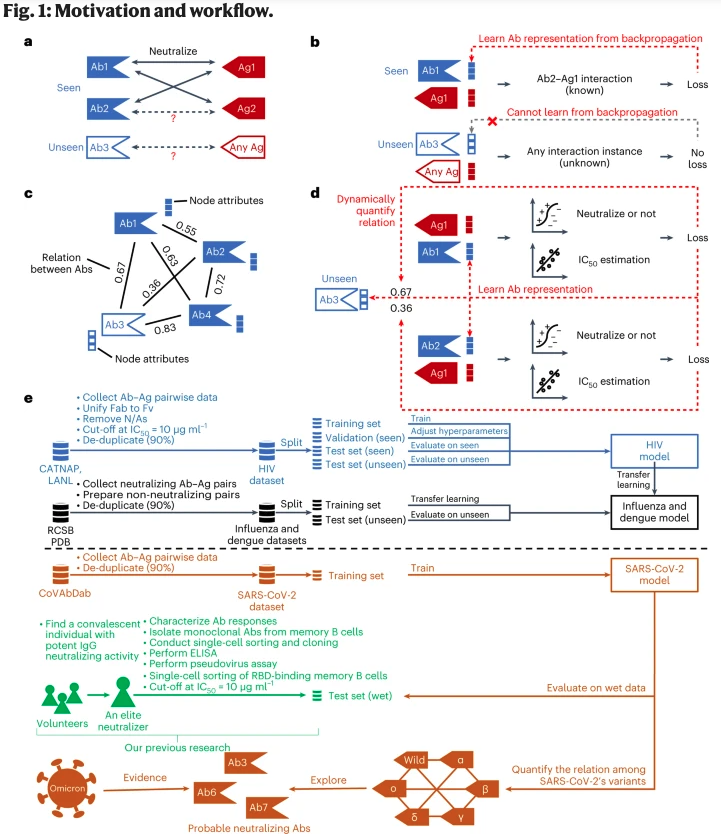
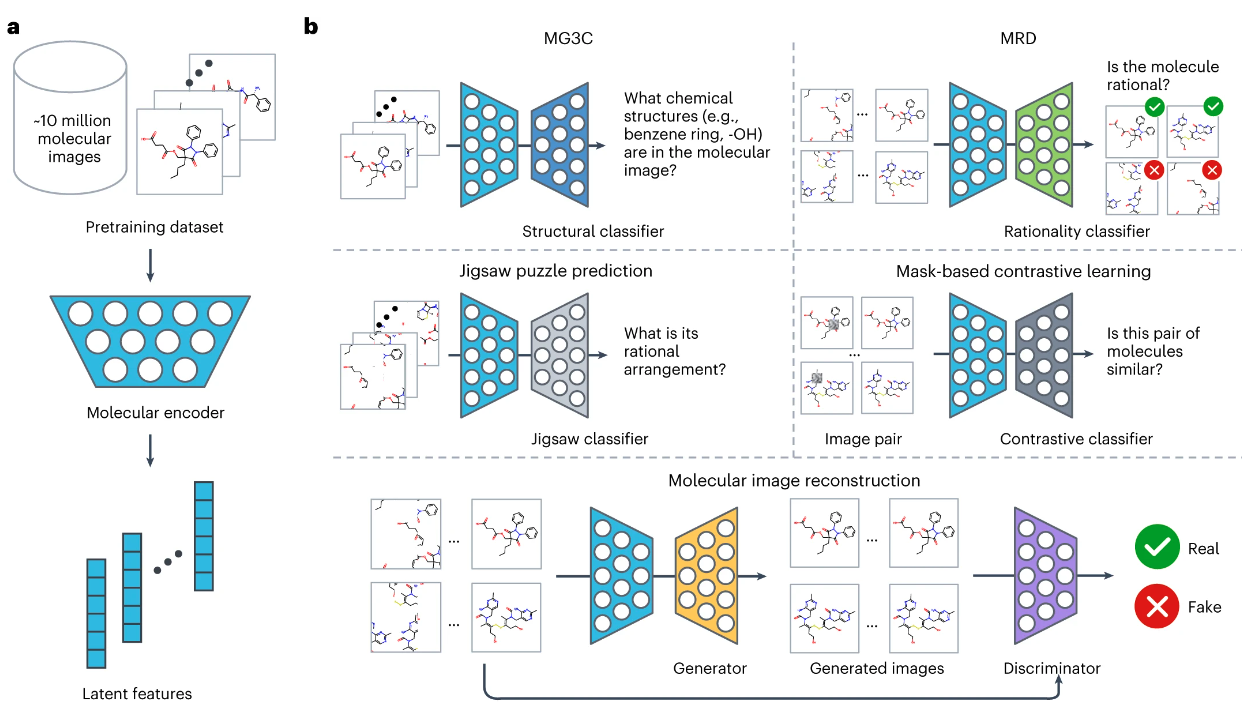
5. 利用自我监督的图像表示学习框架准确预测分子特性和药物目标
题目:Accurate prediction of molecular properties and drug targets using a self-supervised image representation learning framework
杂志:Nature Machine Intelligence
IF: 15
时间:17 November 2022
链接:https://www.nature.com/articles/s42256-022-00557-6
摘要
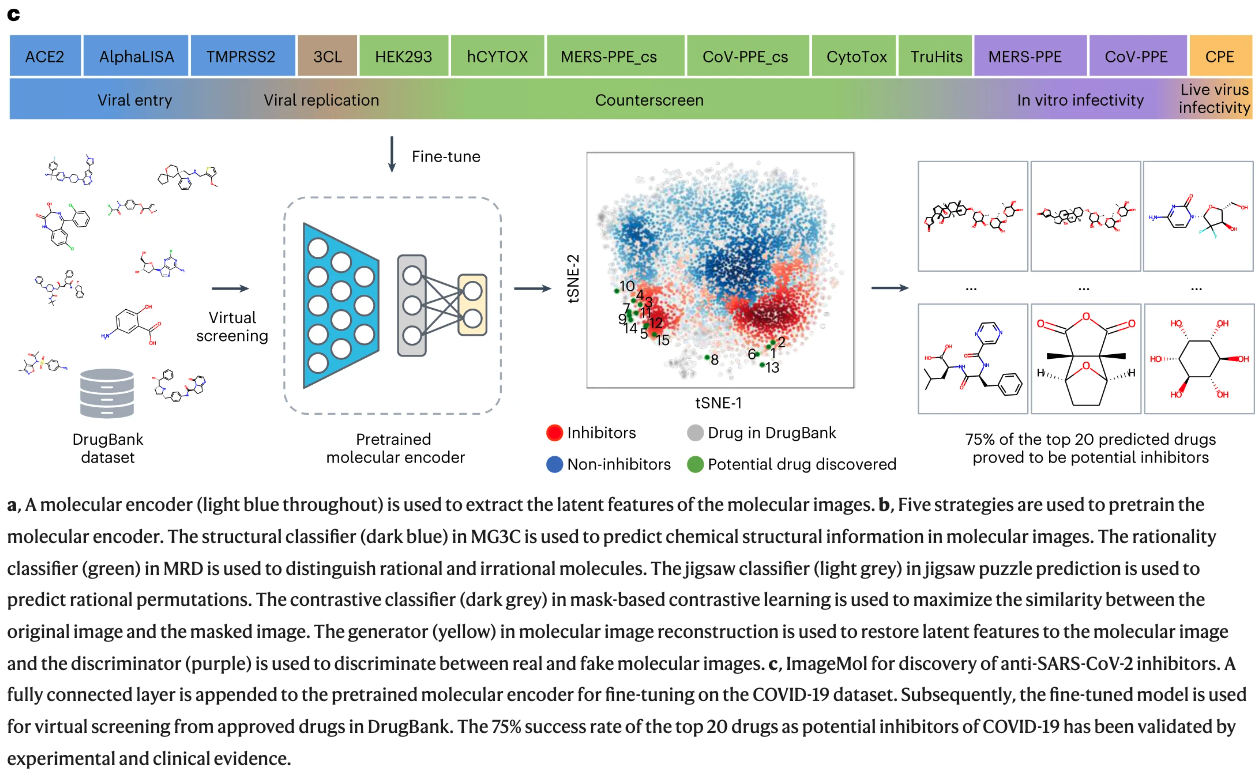
一种药物的临床疗效和安全性是由其在人体中的分子特性和目标决定的。然而,在人类甚至动物模型中对所有化合物进行全蛋白质组评估是具有挑战性的。在这项研究中,我们提出了一个无监督预训练的深度学习框架,名为ImageMol,在1000万个无标签的类药物、生物活性分子上进行预训练,以预测候选化合物的分子目标。ImageMol框架旨在根据像素中分子的局部和整体结构特征,从无标记的分子图像中预训练化学表征。我们证明了ImageMol在评估51个基准数据集的分子特性(即药物的代谢、脑穿透和毒性)和分子靶点概况(即β-分泌酶和激酶)方面的高性能。ImageMol在国家转化科学促进中心的13个高通量实验数据集中显示了识别抗SARS-CoV-2分子的高准确度。通过ImageMol,我们确定了可能用于治疗COVID-19的候选临床3C类蛋白酶抑制剂。