一句话评价
scRNA-seq目前最佳实践分析教程
文章信息
题目:Current best practices in single-cell RNA-seq analysis: a tutorial
杂志:Molecular systems biology
时间:Jul. 13, 2019
链接: doi: http://dx.doi.org/10.1101/701680
figure
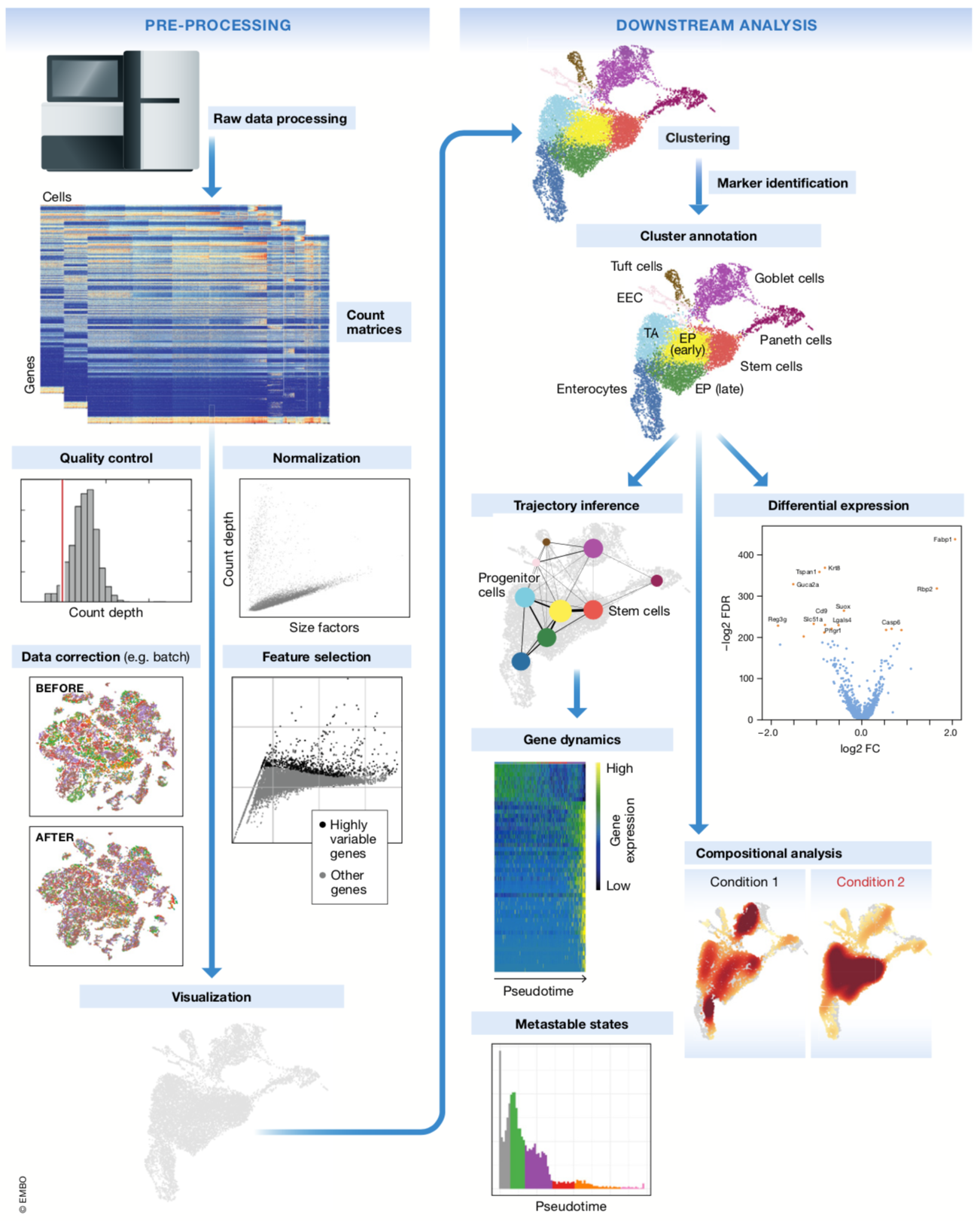
文章介绍
这篇文章通过对单细胞方法的总结,提出了一个目前最佳的实践分析流程,代码在https://github.com/theislab/single-cell-tutorial。
单细胞上游分析:raw data 经过处理得到Count 矩阵,然后进行质量控制,标准化,数据矫正,特征选择和可视化
单细胞下游分析:聚类,marker基因的鉴定,注释,轨迹预测,差异基因,基因调控网络,模块组成等
常见平台:
- 命令行平台:
- Scater
- Seurat
- Scanpy
- 图形用户交互平台:
- Granatum
- ASAP
- FASTGenmics
常用的方法工具:
- Scater: https://bioconductor.org/packages/release/bioc/vignettes/scater/inst/doc/vignette-intro.html
- Seurat:https://satijalab.org/seurat/vignettes.html
- Scanpy: https://scanpy.readthedocs.io/en/stable/tutorials.html
教程:
Analysis of single-cell RNA-seq data with R and Bioconductor
Analysis of single cell RNA-seq data
- Current best-practices in single-cell RNA-seq: a tutorial
碎碎念
方法更新太快,这里推荐的最佳流程不一定在任何时候都适合,还是要根据目的和具体情况选择
每日文献摘要:第6篇 2019年09月20日 周五