01 | 多机构医学成像上的端到端隐私保护的深度学习
文章信息
题目:End-to-end privacy preserving deep learning on multi-institutional medical imaging
杂志:Nature Machine Intelligence
IF: 25.898
时间:24 May 2021
链接:https://www.nature.com/articles/s42256-021-00337-8
摘要
涉及到大型、多国数据集的高性能医学成像人工智能系统需要考虑到数据隐私保护的问题,以便模型可以在不需要数据传输的情况下训练敏感数据。在这里,他们提出了PriMIA(Privacy-preserving Medical Image Analysis,保护隐私的医学图像分析),这是一个免费的开源软件框架,用于在医学成像数据上进行不同程度的隐私、安全聚合的联合学习和加密推理。他们用一个真实的案例研究来测试PriMIA,一个深度卷积神经网络模型可以实现对儿科胸部X光片专家级的分类;所产生的模型的分类性能与本地非安全训练的模型相当。此外,他们从理论上和经验上评估了框架的性能和隐私保证,并证明所提供的保护可以防止基于梯度的模型反转攻击重建可用的数据。最后,他们在一个端到端加密的远程推理场景中成功地使用了训练好的模型,使用安全的多方计算来防止数据和模型的泄露。
背景介绍
高性能人工智能系统的共同点是需要大量和多样化的数据集来训练ML模型,通常通过代表数据所有者的自愿数据共享和多机构或多国的数据集积累来实现。通常情况下,病人数据在来源机构被匿名或假名化,然后被传输并存储在分析和模型训练的地点(被称为集中数据共享)。 然而,事实证明,匿名化对重新识别攻击的保护是不够的。因此,从法律和道德的角度来看,大规模收集、汇总和传输病人数据是至关重要的。此外,控制个人健康数据的存储、传输和使用是患者的一项基本权利。集中的数据共享实际上消除了这种控制,导致了主权的丧失。此外,匿名数据一旦被传输,就不容易被追溯纠正或增加,例如引入可用的额外临床信息。
尽管有这些担忧,对数据驱动的解决方案的需求不断增加,可能会增加与健康有关的数据收集,不仅来自医学成像数据集、临床记录和医院病人数据,而且还包括例如通过可穿戴健康传感器和移动设备。因此,需要创新的解决方案来协调数据和保护隐私。安全和保护隐私的机器学习(Secure and privacy-preserving machine learning,PPML)旨在保护数据安全、隐私和保密性,同时仍然允许从数据中得出有用的结论或用于模型开发。在实践中,尽管本地数据的可用性有限,PPML仍能在低信任度环境中实现最先进的模型开发。这种环境在医学上很常见,数据所有者不能依赖其他方的隐私和保密性。PPML还可以为模型所有者提供保证,即他们的模型不会被修改、窃取或滥用,例如在使用过程中对其进行加密。这为可持续的合作模型开发和商业部署奠定了基础,减轻了对资产保护的担忧。
已有的研究工作
最近的工作显示了PPML在生物医学科学,特别是医学成像方面的效用。例如,联邦学习(federated learning,FL)是一种去中心化的计算技术,它基于将机器学习模型分配给数据所有者(也被称为计算节点)进行分散的训练,而不是集中聚集数据集。它被作为一种促进多国合作的方法提出,同时避免了数据传输。在COVID-19大流行的背景下,FL被用来允许保留数据主权和对数据存储库执行地方治理政策。在医学成像方面,最近的研究表明,深度学习模型的联邦训练在脑瘤分割或乳腺密度分类方面的表现与本地训练不相上下,它促进了来自更多不同来源的数据的纳入,从而提高了泛化能力。然而,FL本身并不是一项完全保护隐私的技术。以前的研究表明,反转攻击可以从模型权重或梯度更新中重建图像,具有令人印象深刻的视觉细节。此外,在推理即服务的设置中,将模型暴露给不信任的第三方可以使模型被滥用或直接被盗。因此,FL必须由额外的隐私增强技术来增强,以真正保护隐私。例如,带有权重或梯度更新的安全聚合(secure aggregation,SecAgg)或差分隐私(differential privacy,DP)的FL可以防止数据集重建攻击,而在模型推理期间利用安全多方计算(secure multi-party computation,SMPC)协议可以保护使用中的模型。我们在以前的工作中对这些技术进行了概述
创新点
提出的PriMIA,是一个免费的、开源的框架,用于医学图像上的端到端隐私保护的分散式深度学习。该框架包含了不同的私有联合模型训练,并对模型更新进行加密聚合,以及加密的远程推理。主要创新点包括:
展示了在临床上具有挑战性的儿科胸部放射学分类任务上,使用PriMIA的隐私增强的FL技术在公共互联网上训练深度卷积神经网络(CNN)。
该框架与广泛的医学成像数据格式兼容,易于用户配置,并引入了FL训练的功能改进(加权梯度下降/联合平均、多样化数据增强、局部提前停止、全联盟超参数优化、差分隐私(differential privacy, DP)数据集统计交换),提高了灵活性、可用性、安全性和性能。
他们考察了使用和不使用隐私增强技术训练的模型的计算和分类性能,与在累积数据集上集中训练的模型、在数据子集上训练的个性化模型以及在未见过的真实数据集上与放射科专家进行对比,以评估医学成像研究中的各种典型场景。
评估了框架的理论和经验上的隐私和安全保证,并提供了在一些训练场景下对模型应用最先进的基于梯度的模型反转攻击的例子。
展示了在安全推理即服务的情况下利用训练好的模型,而不需要披露数据或模型的纯文本,并展示了我们的SMPC协议在推理延迟上的改进。
方法介绍
PriMIA是作为开源PPML工具的PySyft/PyGrid生态系统的延伸而开发的。PySyft(https://github.com/OpenMined/PySyft)是一个Python框架,允许远程执行机器学习任务(例如,张量操作),并通过与PyTorch等常见机器框架的对接,进行加密的深度学习。PyGrid为在服务器和边缘计算设备上部署此类工作流提供了服务器/客户端功能。这些框架所提供的通用功能的详细描述可以在我们之前的工作中找到(A generic framework for privacy preserving deep learning)。PriMIA通过与DICOM等医疗成像数据格式的原生兼容,以及能够对任意模式和维度的医疗数据集(例如,计算机断层扫描、放射成像、超声和磁共振成像)进行操作,将这些功能建立在医学成像特定应用的基础上。在上述PPML技术之外,它还为医学成像分析工作流程中的常见挑战提供了解决方案,如数据集不平衡、高级图像增强、联邦范围的超参数调整功能。此外,它还为从用户机器上的本地实验到远程计算节点上的分布式训练等应用提供了一个可访问的用户界面,以促进PPML最佳实践在医疗联合体中的应用。
源码和数据在:
- https://zenodo.org/record/4545599#.Y7XygezMJhE
- https://github.com/gkaissis/PriMIA
- https://g-k.ai/PriMIA/
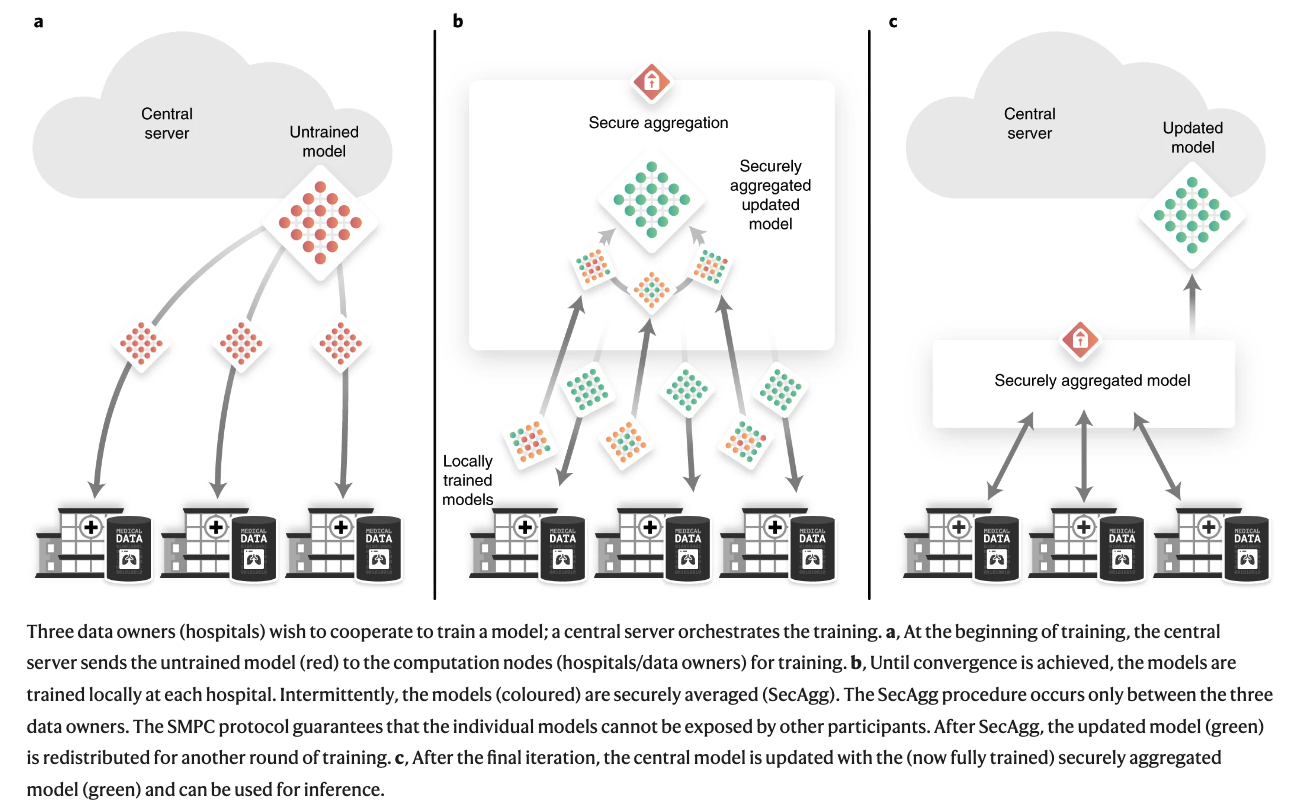
系统架构
三个数据所有者(医院)希望合作训练一个模型;一个中央服务器协调训练。 a, 在训练开始时,中央服务器将未经训练的模型(红色)发送到计算节点(医院/数据所有者)进行训练。 b, 直到达到收敛,模型在每个医院进行本地训练。间断地,模型(彩色)被安全地平均化(SecAgg)。SecAgg程序只发生在三个数据所有者之间。SMPC协议保证单个模型不能被其他参与者暴露。在SecAgg之后,更新的模型(绿色)被重新分配到另一轮训练中。 c, 在最后的迭代之后,中心模型被更新为(现在完全训练好的)安全聚合的模型(绿色),并可用于推理。
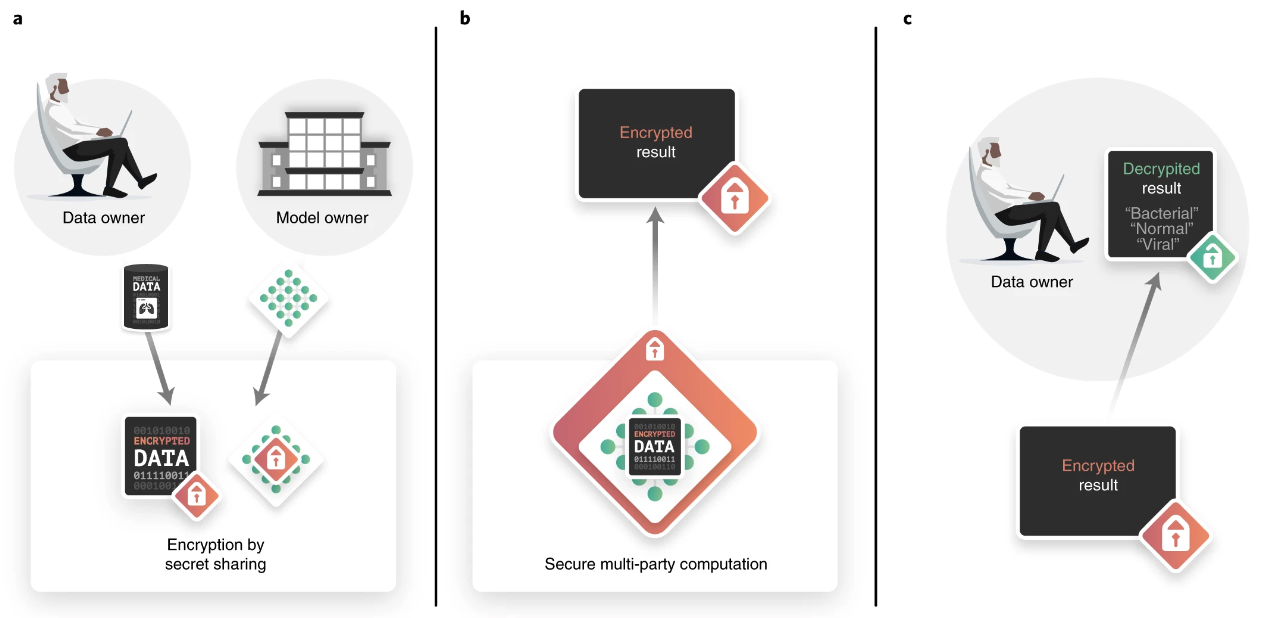
加密推理过程概述
数据所有者(在这种情况下,位于远程地点的医生)通过互联网请求模型的推理结果,但希望他们持有的病人数据保持机密。同样地,模型所有者提供推理服务,但希望对他们的模型保密。使用安全的多方计算(secure multi-party computation, SMPC)可以实现以下情况。 a,最初数据所有者和模型所有者分别使用秘密共享对数据和模型进行加密。这个过程依赖于将数据/模型分割成股份,这些股份本身不包含任何可用的信息,因此可以与另一方交换(共享)。b,然后通过使用SMPC共同计算一个函数(在这种情况下是神经网络推理程序)来进行推理。