一句话评价
肿瘤的分子进化特征为肿瘤的早期诊断提供参考
文章信息
题目:Molecular Evolution of Early-Onset Prostate Cancer Identifies Molecular Risk Markers and Clinical Trajectories
杂志:Cancer Cell
时间:December 10, 2018
链接: https://doi.org/10.1016/j.ccell.2018.10.016
figure
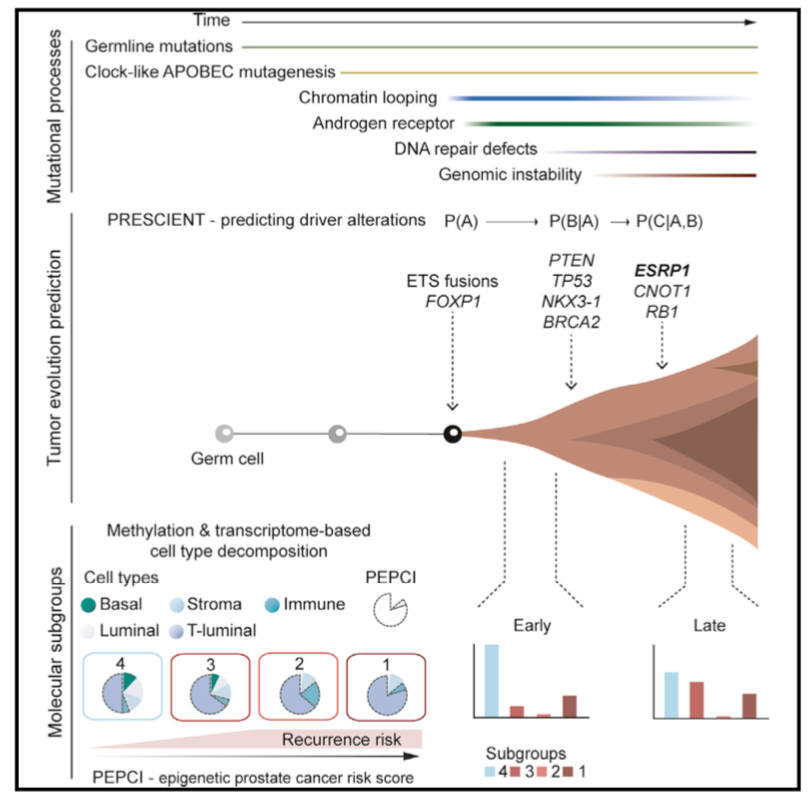
文章介绍:
这篇文章发表于2018年12月,由丹麦哥本哈根大学和德国海德堡大学主导完成,文章利用前列腺癌早期的分子进化特征鉴定前列腺癌不同等级风险的标志物和临床发展轨迹,主要结果包括4个方面:
- APOBEC3的类时钟(clock-like)突变过程介导了前列腺癌(Prostate Cancer,PC)的早期突变
- 鉴定出能够区分中等危险疾病(intermediate-risk)的四个分子亚群
- ESRP1位点的重排与癌症的侵袭性和增殖性相关
- 开发了利用DNA测序数据预测PC临床轨迹的方法
文中用到的数据包括WGS,450K 甲基化以及RNA-seq共292个前列腺癌样本,当然不同数据类型的样本并不是一一对应的,另外还用到了公共的Hi-C数据(具体数据来源在文中还没有找到)。
注:EOPC(early-onset PC): 早期前列腺癌; LOPC(late-onset PC): 晚期前列腺癌
| 数据类型 | EOPC 样本数 | LOPC样本数 |
|---|---|---|
| WGS | 184 | 85 |
| Methylomes(450K) | 203 | 45 |
| mRNA-seq | 96 | |
| Hi-C data |
作者利用这些数据进行了什么分析呢
首先鉴定EOPC的体细胞突变模式
通过somatic和germline的call变异流程,分析SNVs、InDels和SVs。 从SNVs的结果来看,与以前研究的结果一致,前列腺癌的SNVs非常低;EOPC与LOPC相比,EOPC的SNVs数量低于LOPC(平均每M b的SNVs数量,EOPC=0.47,LOPC=0.53),其中EOPC中TP53的变异率最高。SVs是前列腺癌出现频率较高的变异,SVs常与PC中肿瘤抑制基因的反复融合的形成或丢失有关。基于此作者鉴定了复发性基因组变异位点(recurrent genomic altered loci ,RGA),结果揭示70%的EOPC携带的SV与ETS融合基因的形成有关,EOPC中第二、三易变异的位点分别是8号染色体的NKX3-1和3号染色体的FOXP1基因附近。为了鉴定RGAs与年龄的关系(即RGAs与前列腺癌发展的早期阶段和晚期阶段的关联),作者继续对LOPC做了对应的基因组变异分析,结果显示LOPC中RGAs的整体比例高于EOPC,另外EOPC中展现出较高的单克隆结构,表明EOPC可能主要与克隆起源有关。
肿瘤发展的过程常伴随这表观结构的变异,如DNA双链的开放和紧密压缩状态的改变。实验室之前的研究发现EOPC基因组中断裂点(breakpoits)常出现在雄性激素受体结合位点(AR-binding sites)附近,这增加了年龄相关的染色质状态改变影响断点发生的可能性。因此,他们整合公共的Hi-C数据检测与特定染色质区域相关的EOPC基因组断裂点,发现断裂点与染色质loop和H3K27ac peaks的数量显著相关。表明EOPC的断裂点主要出现在染色质开放区域,与活性元件相关;LOPC断裂点主要出现在异染色质区域。
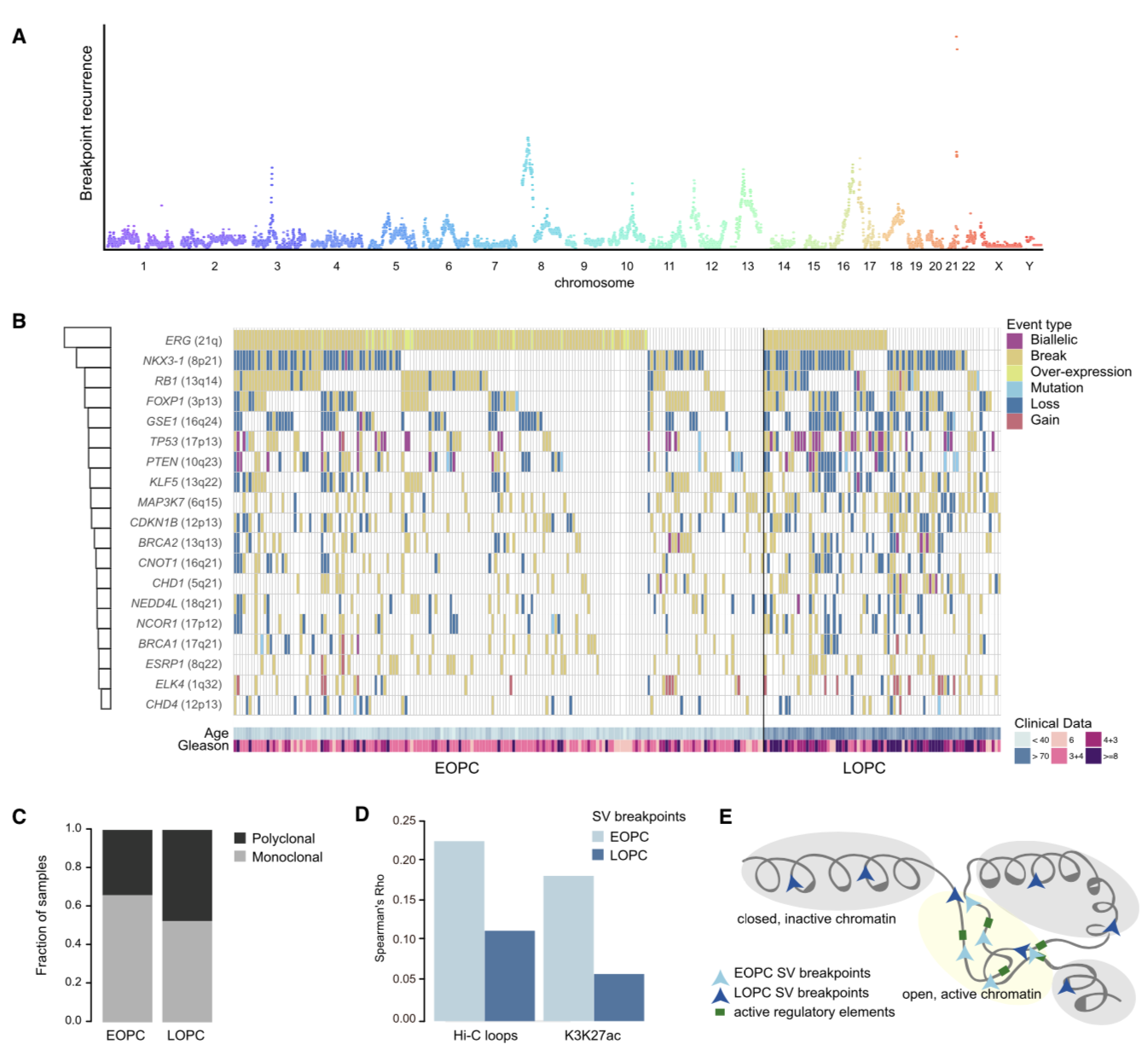
小结:这一部分主要利用基因组数据分析前列腺癌早期和晚期的变异特征(SNVs,InDels和SVs)。展示了EOPC和LOPC中断裂点的不同的分布特征,以及与染色质状态的变化。
疑问:有两个概念不怎么理解,一个是RGA,一个是breakpoints,而这两个概念又是这篇文中分析的重点内容。
- RGAs(recurrent genomic altered loci ): 字面意思反复出现的基因组变异位点,文中说SVs常与PC中肿瘤抑制基因的反复融合的形成或丢失有关,基于此作者鉴定了RGAs。RGAs是否是染色质结构变异中的DNA重排呢?与SV以及融合基因有什么关联?如何鉴定RGAs呢?
- Genome Breakpoints:基因组断裂点。B图(瀑布图)中对变异类型的描述中有个break,可能和Genome Breakpoints有关,是一个变异类型?那么和loss, gain,mutation有什么区别呢?查阅资料发现breakpoints结各种结构变异类型分析中的一个概念
- 另外文中说做了germline和somatic的call变异,但只提供了WGS数据,为什么没有WES数据?只用WGS进行的somatic variants的分析吗?
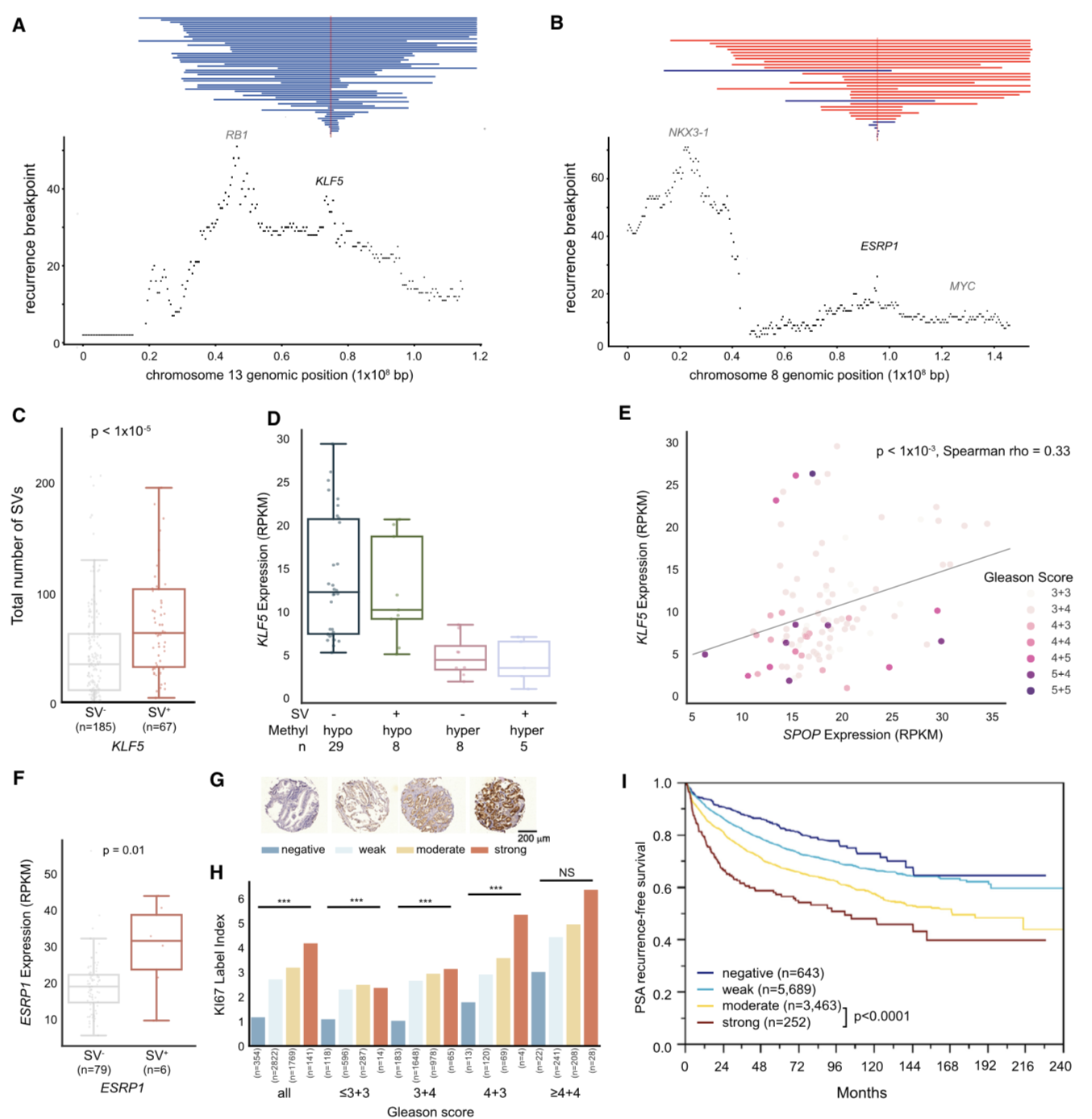
DNA重排分析
DNA重排是结构变异的一个形式。作者鉴定到EOPC中有两个RGAs位于13q22和8q22。 13q22的最小重叠峰区域以KLF5为中心,以往的研究表明KLF5编码一个参与抑制细胞增殖的转录激活因子, 13q22的丢失与KLF5 mRNA水平的降低以及SV和SNV负荷的整体增加有关。该研究发现KLF5与SPOP(有一个鼠的胚胎干细胞研究中发现SPOP是KLF5的一个靶基因,是一个泛素连接酶基因) mRNA表达水平呈正相关关系,但是与SPOP的突变无关。
8q22处的一个区域显示以ESRP1为中心的反复基因组复制,最小重叠峰区域在MYC附近。这些重复与ESRP1和MYC都有重叠,但只有ESRP1在PC样本中显示出显著的mRNA水平增加。此外,ESRP1重排与格里森评分(GS,前列腺癌评估的一个重要指标)升高显著相关。ESRP1蛋白水平的升高与增殖率升高相关。另外发现其可以作为一个独立的预后标志物。
小结: 根据DNA重排分析,发现ESRP1重排与ESRP1蛋白表达增加、细胞增殖水平升高、GS和肿瘤分期升高有关,并证明ESRP1表达是PC中一个独立的预后生物标记物。
基因组变异的背景知识
- 单碱基变异,即单核苷酸多态性(SNP),最常见也最简单的一种基因组变异形式;
- 很短的Insertion 和 Deletion,也常被我们合并起来称为Indel。主要指在基因组某个位置上发生较短长度的线性片段插入或者删除的现象。强调线性的原因是,这里的插入和删除是有前后顺序的与下述的结构性变异不同。Indel长度通常在50bp以下,更多时候甚至是不超过10bp,这个长度范围内的序列变化可以通过Smith-Waterman 的局部比对算法来准确获得,并且也能够在目前短读长的测序数据中较好地检测出来
- 基因组结构性变异(Structure Variantions,简称SVs),这篇文章的重点,通常就是指基因组上大长度的序列变化和位置关系变化。类型很多,包括长度在50bp以上的长片段序列插入或者删除(Big Indel)、串联重复(Tandem repeate)、染色体倒位(Inversion)、染色体内部或染色体之间的序列易位(Translocation)、拷贝数变异(CNV)以及形式更为复杂的嵌合性变异。
参考来源:碱基矿工黄树嘉老师的文章—https://zhuanlan.zhihu.com/p/40290546
经典的SVs综述:
- Alkan, C., Coe, B. P. & Eichler, E. E. Genome structural variation discovery and genotyping. Nature reviews. Genetics**12**, 363–76.
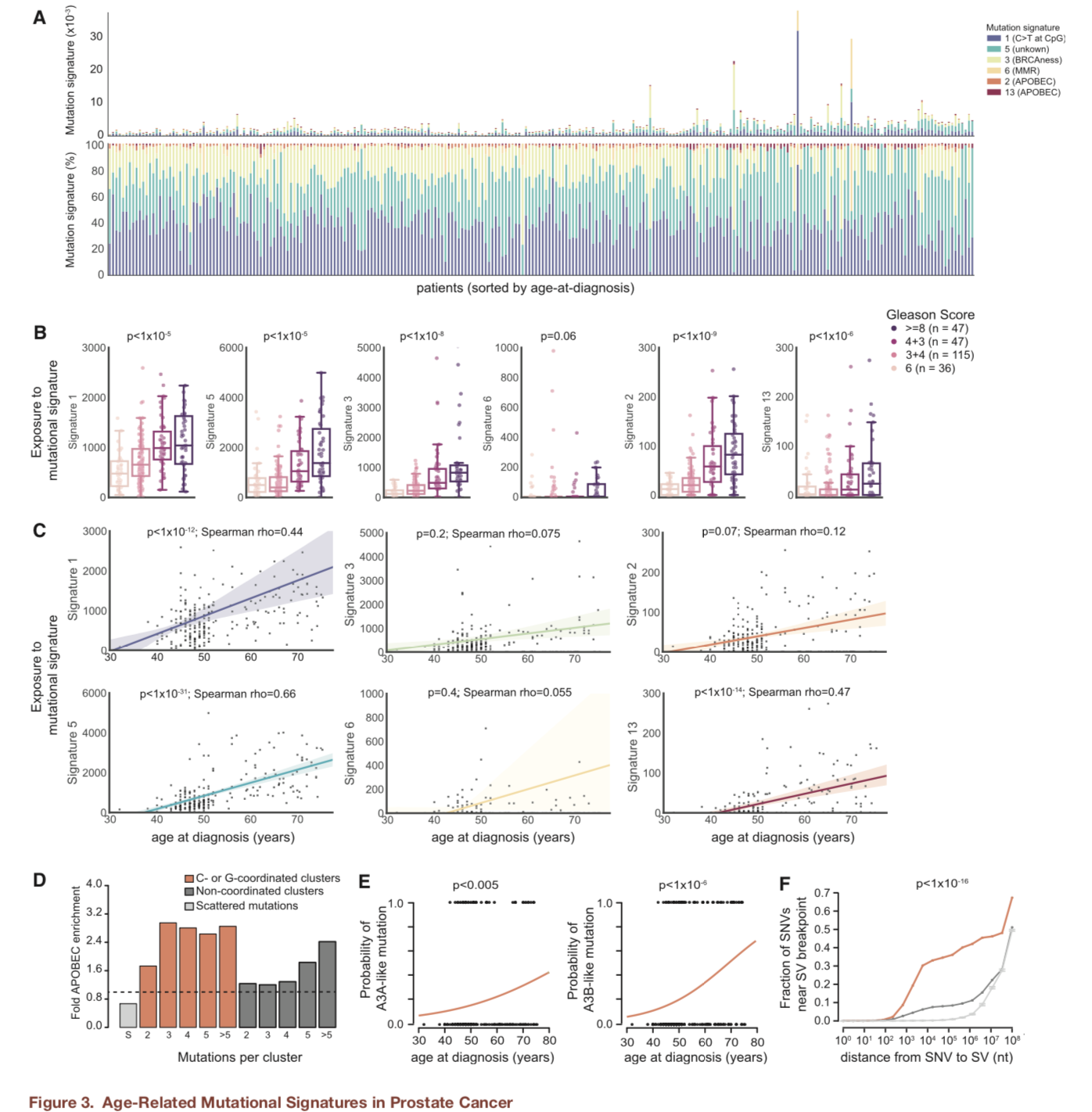
突变特征(mutational signature)分析
突变信号(mutational signature)可以用来描述作用于肿瘤细胞的内源性和外源性介导的突变过程。通过突变特征的分析发现APOBEC3的类时钟(clock-like)突变过程介导了PC的早期突变,APOBEC3蛋白是一种胞苷脱氨酶,在单链DNA复制周期中起到限制反转录因子的作用,但也可以诱导癌症基因组的突变。
背景知识
mutaional signature: 下文内容来自:作者-ksxg ,公众号BioKSXG,文章题目:看文献,详解Mutational signatures(突变指标/签名/特征)
不同的体细胞突变过程会产生各种结果,不同的突变过程产生突变类型的独特组合,称为“Mutational Signatures”。截止到2018年已经有30种定义的Mutational Signatures:
Signature 涉及的癌症种类 病因学 额外的突变特征 Comments 1 所有癌症,大部分样品 5-甲基胞嘧啶脱氨基作用自发的内源性突变过程导致 小数量的小插入和在大多数组织类型的删除 与诊断年龄相关 2 主要在子宫癌和膀胱癌 AID/APOBEC家族的胞嘧啶核苷脱氨酶的活动 观察到外显子中转录链偏差的突变,但是在内含子中不存在或者较弱 2和13常在同一样本中被鉴定出,常伴随kataegis突变机制 3 乳腺癌、卵巢癌、胰腺癌 通过同源重组的DNA双链断裂修复 断点连接中活跃的大片段(大于3bp)插入和删除,伴随着重叠的微同源序列 乳腺癌、胰腺癌和卵巢癌中生殖系和体细胞的BRCA1和BRCA2突变,特别是胰腺癌中铂疗法 4 头颈癌、肝癌、肺腺癌、肺鳞癌、小细胞肺癌、食道癌 吸烟相关,烟草诱变剂 偏向C >A突变,也与CC > AA二核苷酸替换相关 29也与烟草咀嚼相关癌症有关,但是与4不同 5 所有癌症,大部分样品 未知 T > C替换(ApTpN) 无 6 17种癌症,但主要在结直肠和子宫癌症 DNA错配修复缺陷和微卫星不稳定的肿瘤 单核/多核苷酸重复中活跃的小片段(小于3bp)的插入和删除 4和15、20、26属于DNA错配修复缺陷相关的四大mutational signature 7 皮肤癌,头部和口腔鳞状上皮癌 紫外线暴露 大量的CC > TT二核苷酸突变,被转录的核苷酸切除修复嘧啶突变 无 8 乳腺癌和成神经管细胞瘤 微弱的C >A替换,与双核苷酸替换,尤其是CC > AA相关 未知 无 9 慢性淋巴细胞白血病和恶性B细胞淋巴瘤 聚合酶η引起的突变模式,涉及体细胞超突变的活动 未知 9中,与那些没有超突变的免疫球蛋白基因相比,具有免疫球蛋白基因超突变(IGHV-mutated)的慢性淋巴细胞白血病有大量突变 10 六种癌症,尤其是肠癌和子宫癌,通常会在一小部分样品中产生大量的突变 容易出错的聚合酶极的活动,10中有大量的复发性杆体细胞突变,即Pro286Arg Val411Leu C >A突变(TpCpT)和T > G突变(TpTpT) 与大部分突变的癌症样本相关,这个mutational signature定义为ultra-hypermutators(超-超突变子?不懂) 11 黑色素瘤和胶质母细胞瘤 类似于烷化剂的突变模式 强烈的C > T替换,能被转录的核苷酸切除有效修复 无 12 肝癌 未知 强烈的T > C替换 12在肝癌样本中突变率较小(<20%) 13 22种癌症中,常见于宫颈和膀胱癌 AID/ APOBEC家族的胞嘧啶核苷脱氨酶将胞嘧啶转化为尿嘧啶活动引起的,尤其是APOBEC1, APOBEC3A 和/或者 APOBEC3B,13导致显著的C > G突变 观察到外显子中转录链偏差的突变,但是在内含子中不存在或者较弱 经常和2在同一样品中被鉴定出,病毒感染、逆转录转座子跳跃或组织炎症引起的AID/ APOBEC胞嘧啶核苷脱氨酶激活,常伴随kataegis突变机制 14 四种子宫癌症和一种成人轻度神经胶质瘤样本 未知 未知 在所有样本中都有较高的体细胞突变数(每MB大于200个突变) 15 几种胃癌症和一种小细胞肺癌 DNA错配修复缺陷相关 单核/多核苷酸重复中活跃的小片段(小于3bp)的插入和删除 15和6、20、26属于DNA错配修复缺陷相关的四大mutational signature 16 肝癌 未知 强烈的T > C替换 无 17 食管肿瘤、乳腺癌、肝癌、肺腺癌,B细胞淋巴瘤、胃癌和黑色素瘤 未知 未知 无 18 神经母细胞瘤。此外还在乳腺癌和胃癌癌被观察到 未知 未知 无 19 只在纤维状细胞的星形细胞瘤 未知 未知 无 20 胃和乳腺癌 DNA错配修复缺陷相关 单核/多核苷酸重复中活跃的小片段(小于3bp)的插入和删除 20和6、15、26属于DNA错配修复缺陷相关的四大mutational signature 21 只在胃癌 未知 未知 只在来自同一测序中心的四个样本中被发现,与26有一些类似,只在有15和20的样本中被发现,可能与微卫星不稳定的肿瘤相关 22 肾盂移行细胞癌 发现于马兜铃酸癌症样本 活跃的T>A突变,表明腺嘌呤损伤被转录的核苷酸切除修复 移行细胞癌中突变很高,但在肝癌很低 23 仅在一例肝癌样品中 未知 强烈的C > T替换 无 24 肝癌的子集 黄曲霉毒素暴露 活跃的C >A突变,表明鸟嘌呤损伤被转录的核苷酸切除修复 无 25 霍奇金淋巴瘤 未知 活跃的T>A突变 仅在何杰金氏细胞系中被鉴定出,没有原发霍奇金淋巴瘤的数据 26 乳腺癌、宫颈癌、胃癌、子宫癌 DNA错配修复缺陷相关 单核/多核苷酸重复中活跃的小片段(小于3bp)的插入和删除 26和6、15、20属于DNA错配修复缺陷相关的四大mutational signature 27 肾透明细胞癌的子集 未知 活跃的T>A突变,断点连接中活跃的大片段(大于3bp)插入和删除,伴随着重叠的微同源序列 无 28 胃癌的子集 未知 未知 无 29 仅在齿龈颊口腔鳞状细胞癌 烟草咀嚼习惯相关的样本 偏向C >A突变,也与CC > AA二核苷酸替换相关 4也与烟草咀嚼相关癌症有关,但是与29不同 30 乳腺癌一小部分子集 未知 未知 无
肿瘤纯度与甲基化联合分析
正常人前列腺组织由基底细胞、管腔细胞和基质细胞组成,而PC则失去基底细胞,获得肿瘤特异性管腔(T-管腔)细胞和浸润性免疫细胞。考虑到DNA甲基化图谱是细胞类型(cell type,ct)特异性的,作者试图通过使用可用的参考甲基化图谱来解释甲基化分析中ct组成的差异。为此,他们从良性前列腺增生患者和前列腺癌患者中获取了额外的切除样本,并进行荧光激活细胞分类,以识别前列腺癌中存在的主要ct,这使他们能够识别前列腺癌基因组中每个甲基化位点的ct特征。
他们发现在高GS肿瘤中,基底细胞和管腔细胞向T管腔细胞和浸润性免疫细胞的反复转移。鉴于T-管腔和免疫细胞含量在鉴别高级别肿瘤中的相关性,他们将此信息合并为前列腺癌肿瘤侵袭性的经纯度调整的表观遗传指数(PEPCI)。他们发现高PEPCI与高pt、高GS和BCR风险增高密切相关。此外,PEPCI能够对中等风险病例进行分层,并且他们在主要LOPC样本的TCGA队列中验证了这一点。最后,我们的PEPCI评分也能够独立预测GS和BCR。
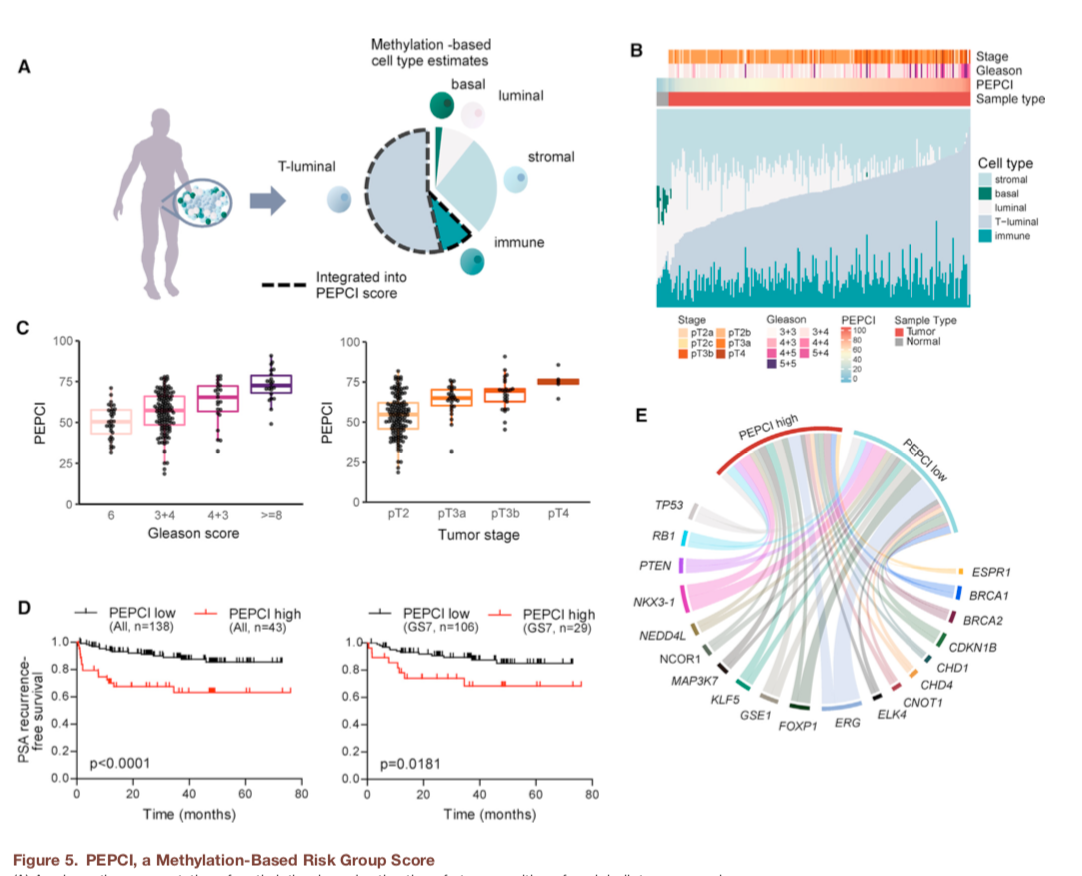
小结:将肿瘤纯度分析与甲基化分析结合分析,提出经纯度调整的表观遗传指数值(PEPCI),证明了PEPCI可以区别不同等级风险PC。
需补充的知识:
- 肿瘤纯度的分析
- 这篇文章提出的PEPCI
整合分析鉴定肿瘤发展的分子亚型
他们利用CLICK算法对96个病人的mRNA-seq数据进行分析,将共表达基因共分成7个不同的CLICK clusters(CC)。然后整合CC表达谱与ct比例以及PEPCI的信息的综合分析将患者分为四个与预后相关的亚组,其在生物学通路的表达上有明显差异。
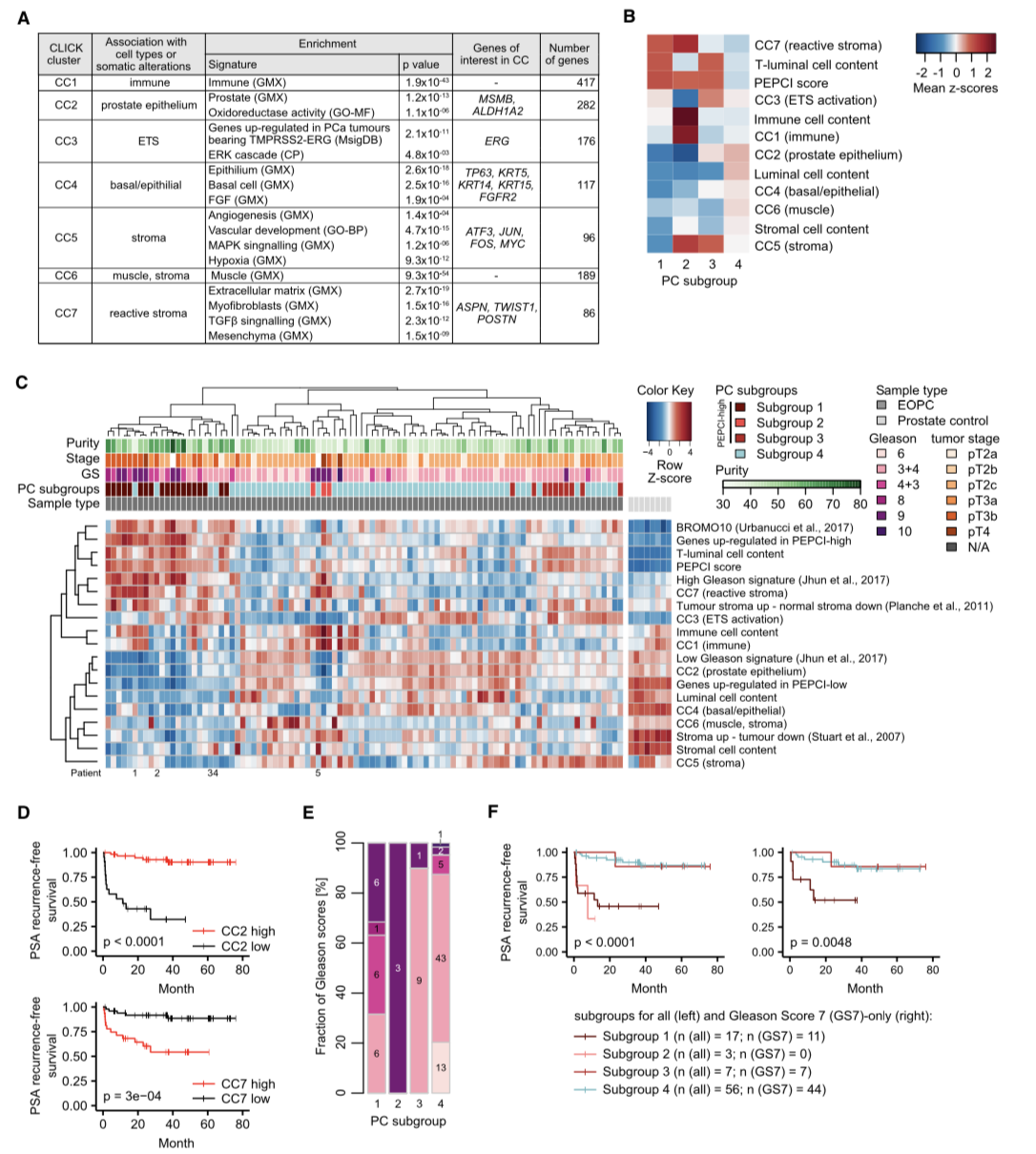
需补充的知识:
- CLICK的聚类方法
预测肿瘤演化顺序的计算方法
他们开发了一种基于条件概率的网络模型PRESCIENT (prediction of sequential changes in the evolution of nascent tumors——预测新生肿瘤演化的顺序变化),用于预测PC和相关临床outcom中体细胞事件的时间序列。
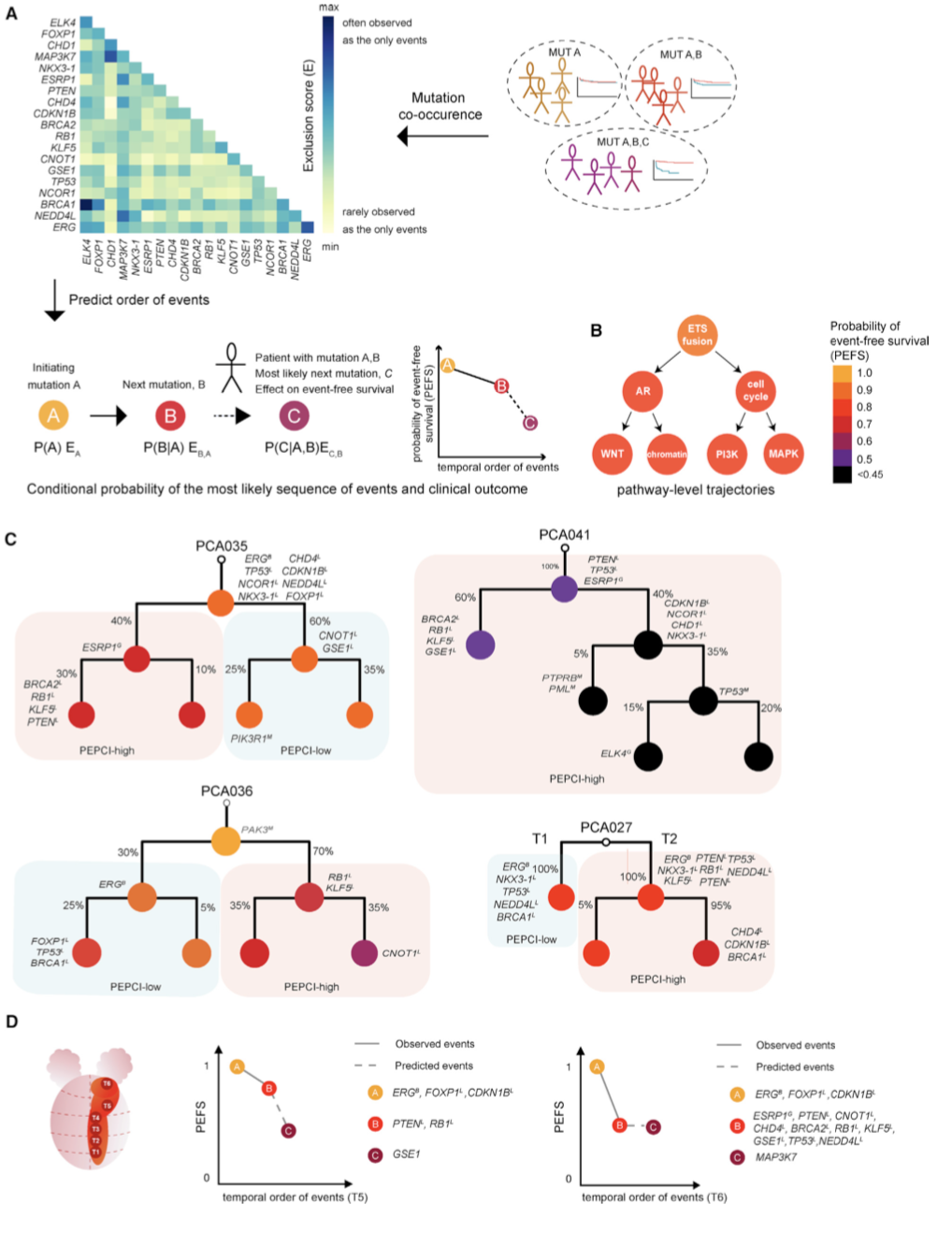
需补充的知识:理解PRESCIENT模型
碎碎念
最后再对这篇文章做一个总结:
这篇文章通过292个前列腺癌样本的WGS,450K 甲基化以及RNA-seq数据以及公共的Hi-C数据,发现早期前列腺癌的分子进化特征可以鉴定前列腺癌不同等级风险的标志物和临床发展轨迹。1)文中首先利用基因组数据分析前列腺癌早期和晚期的变异特征(包括SNVs,InDels和SVs),展示了EOPC和LOPC中断裂点的不同分布特征,以及与染色质状态的变化。2)由于SVs是前列腺癌出现频率较高的变异,所以文中主要做的是SVs相关的分析。基因组重排的分析主要结果是发现了ESRP1位点的重排与癌症的侵袭性和增殖性相关。3)mutational signature的分析发现APOBEC3的类时钟(clock-like)突变过程介导了前列腺癌(Prostate Cancer,PC)的早期突变。4)将肿瘤纯度分析与甲基化分析结合,提出经纯度调整的表观遗传指数值的方法(PEPCI),证明了PEPCI可以区别不同等级风险PC。5)利用CLICK算法对96个病人的mRNA-seq数据进行共表达基因聚类分析,然后整合细胞纯度分析以及PEPCI的信息,将患者分为四个与预后相关的亚组,其在生物学通路的表达上有明显差异。6)开发了一种基于条件概率的网络模型PRESCIENT (prediction of sequential changes in the evolution of nascent tumors——预测新生肿瘤演化的顺序变化),用于预测PC和相关临床outcom中体细胞事件的时间序列。
开发的预测新生肿瘤演化顺序的方法——PRESCIENT :
总之,这篇文章可以作为对肿瘤分子进化特征与肿瘤早期诊断研究感兴趣的参考,文中涉及的方法和开发的新方法也值得学习。
断断续续花了两天时间才看懂了这篇文章的思路框架,方法部分还没有认真细看。粗略估计看懂这篇cancer cell的文章,包括文章思路和结果以及方法,可能需要一个星期的时间。另外一个对我来说这篇文章难于理解的原因是背景知识的缺乏,如结构变异(SVs)的理解,分析原理和方法,以及肿瘤相关方面的背景知识缺乏,我之前是不知道mutaion signature的,也不知道肿瘤中常见的肿瘤纯度,肿瘤进化等是如何分析的,虽然听说过这几个概念,也常见这样的帖子,但是都没有细看。不过认真看一篇文章,学到的知识点真多,以后再看相关的其他文章就有了背景知识。
方法学下次更新
> 每日文献摘要:第11篇 2019年10月26日 周六