Commun. Biol. | Review | 人工智能加速抗生素发现
题目:Accelerating antibiotic discovery through artificial intelligence
杂志:Communication Biology
IF: 6.268
时间:09 September 2021
链接:https://doi.org/10.1038/s42003-021-02586-0
摘要
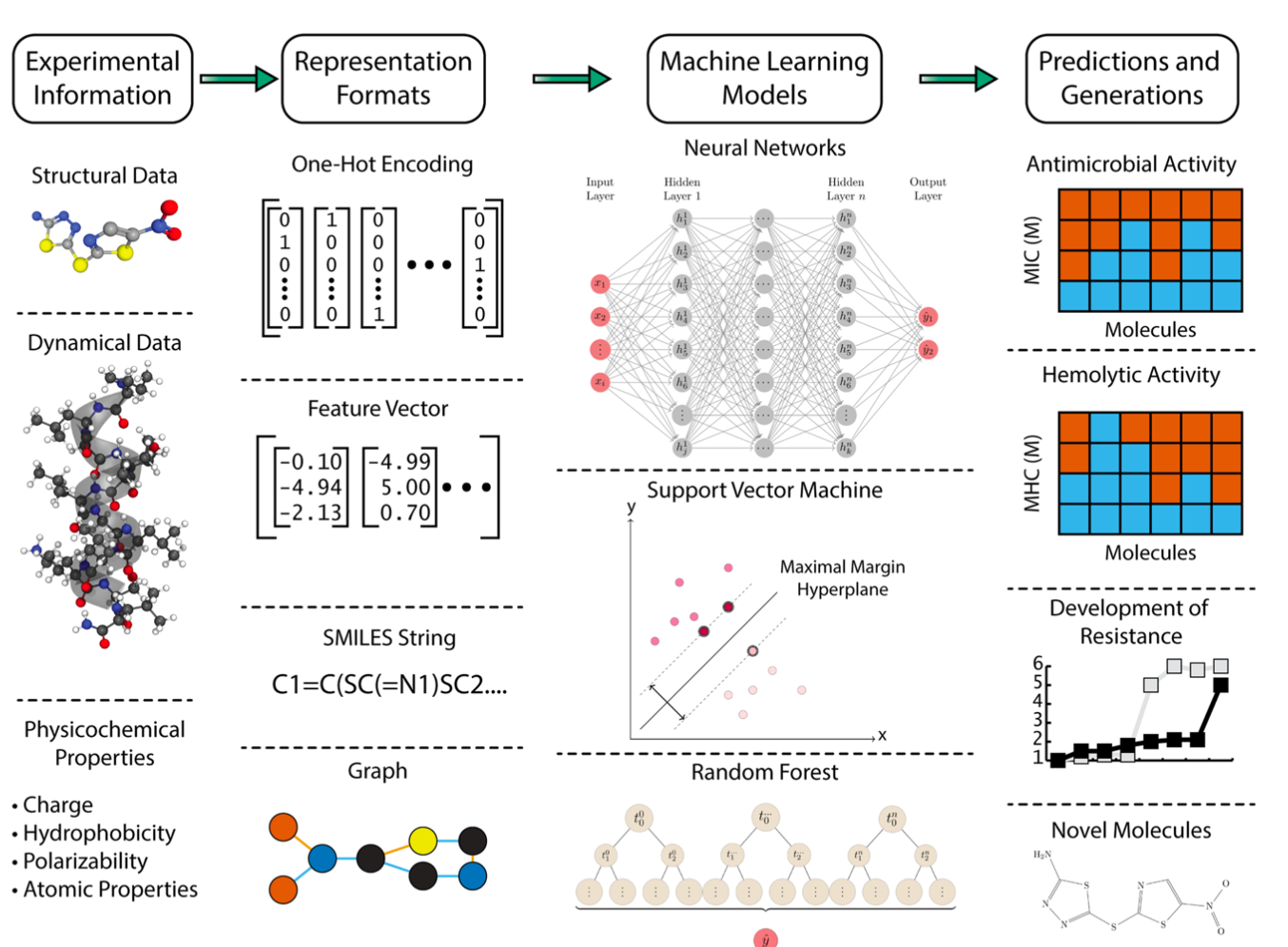
通过靶向结合入侵的生物体,抗生素将自己插入宿主-病原体进化军备竞赛的古老斗争中。随着病原体进化出躲避抗生素的策略,治疗的疗效也逐渐下降,而且必须被取代,这使抗生素与大多数其他形式的药物开发不同。再加上缓慢而昂贵的抗生素开发管道,耐药性病原体的扩散促使人们对有望加快候选药物发现的计算方法产生迫切的兴趣。人工智能(AI)的不断发展鼓励其应用于计算机辅助药物设计的多个层面,并越来越多地应用于抗生素发现。
这篇综述描述了
1)人工智能在发现小分子抗生素和抗菌肽方面的进展。
2)除了对抗菌活性的基本预测外,还强调了抗菌化合物的代表性、药物相似性特征的确定、抗菌剂的抗性和新分子设计。
3)分析了人工智能驱动的抗生素发现中对开放科学最佳实践的吸收情况,并主张将开放性和可重复性作为加速临床前研究的手段。
4)最后,讨论了文献中的趋势和未来探索的领域。
抗生素发现的相关数据库

计算方法发现抗生素的流程
Brief. Bioinformatics |深度神经网络的基因连接矩阵对三阴性乳腺癌亚型分类和基因选择
题目:Classification and gene selection of triple-negative breast cancer subtype embedding gene connectivity matrix in deep neural network
杂志:Briefings in Bioinformatics
IF: 11.62
时间:05 September 2021
链接:https://doi.org/10.1093/bib/bbaa395
摘要
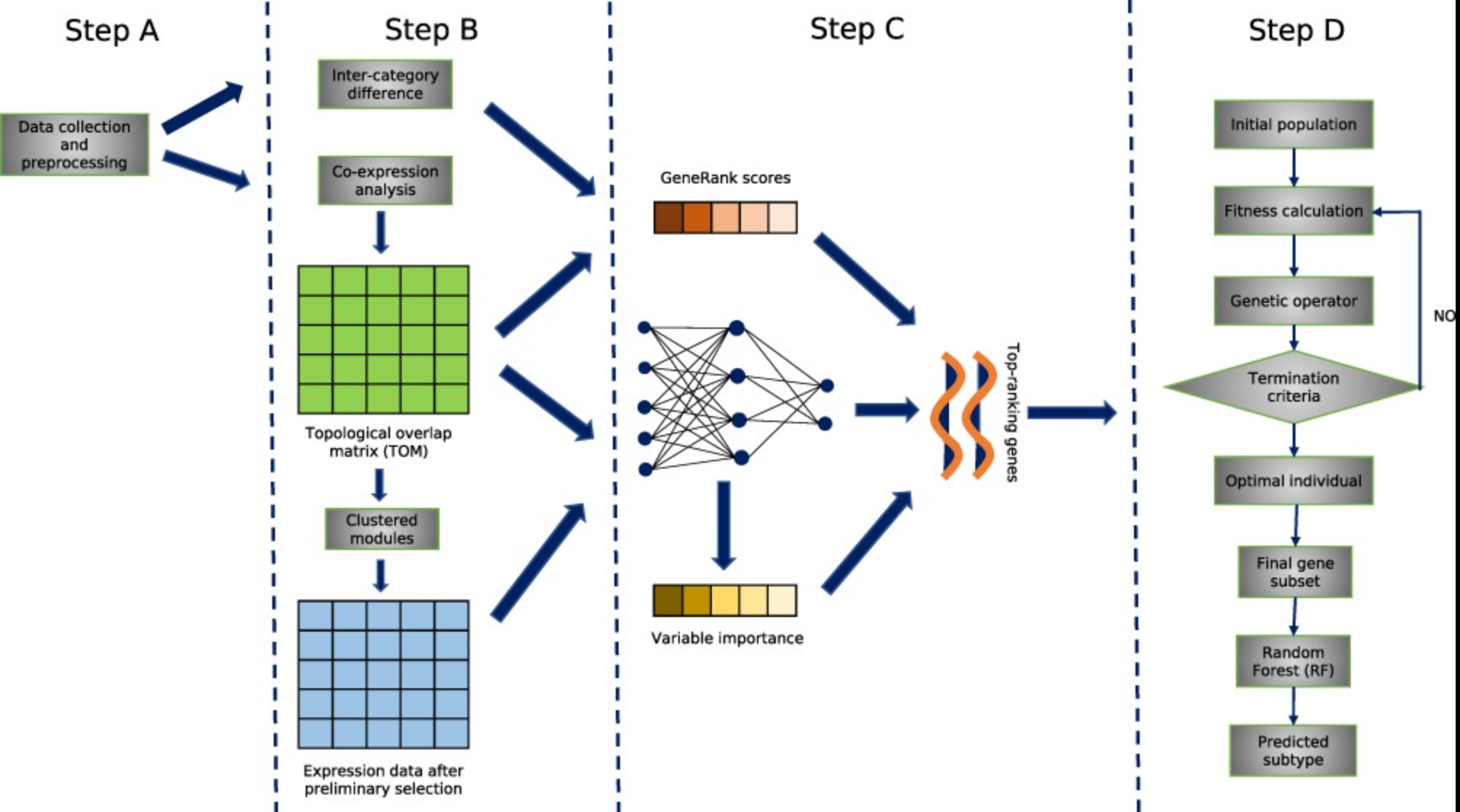
三阴性乳腺癌(TNBC)一直是肿瘤学治疗中具有挑战性的乳腺癌亚型。通常情况下,它可以被划分为不同的分子亚型。对这六种亚型进行准确和稳定的分类对于TNBC的个性化治疗至关重要。在这项研究中,作者提出了一个新的框架来区分TNBC的六个亚型,这也是少数几个基于mRNA和长非编码RNA表达数据 完成分类的研究之一。特别是,他们开发了一种名为DGGA的基因选择方法,该方法在衡量基因重要性的过程中考虑了基因之间的相关信息(WGCNA),然后有效地去除多余的基因。他们提出了一种基因评分方法,将GeneRank分数与深度神经网络(DNN)产生的基因重要性结合起来,考虑到亚型间的鉴别和基因内部的相关性,以提高基因选择性能。更重要的是,他们在DNN中嵌入了基因连接矩阵进行稀疏学习,在获得每个基因的相对重要性的测量时,会额外考虑训练过程中的权重变化。最后,遗传算法被用来模拟自然进化过程,以寻找TNBC亚型分类的最佳子集。他们通过交叉验证来验证所提出的方法,结果表明它可以使用较少的基因来获得更准确的分类结果。
代码在:https://github.com/RanSuLab/TNBC。
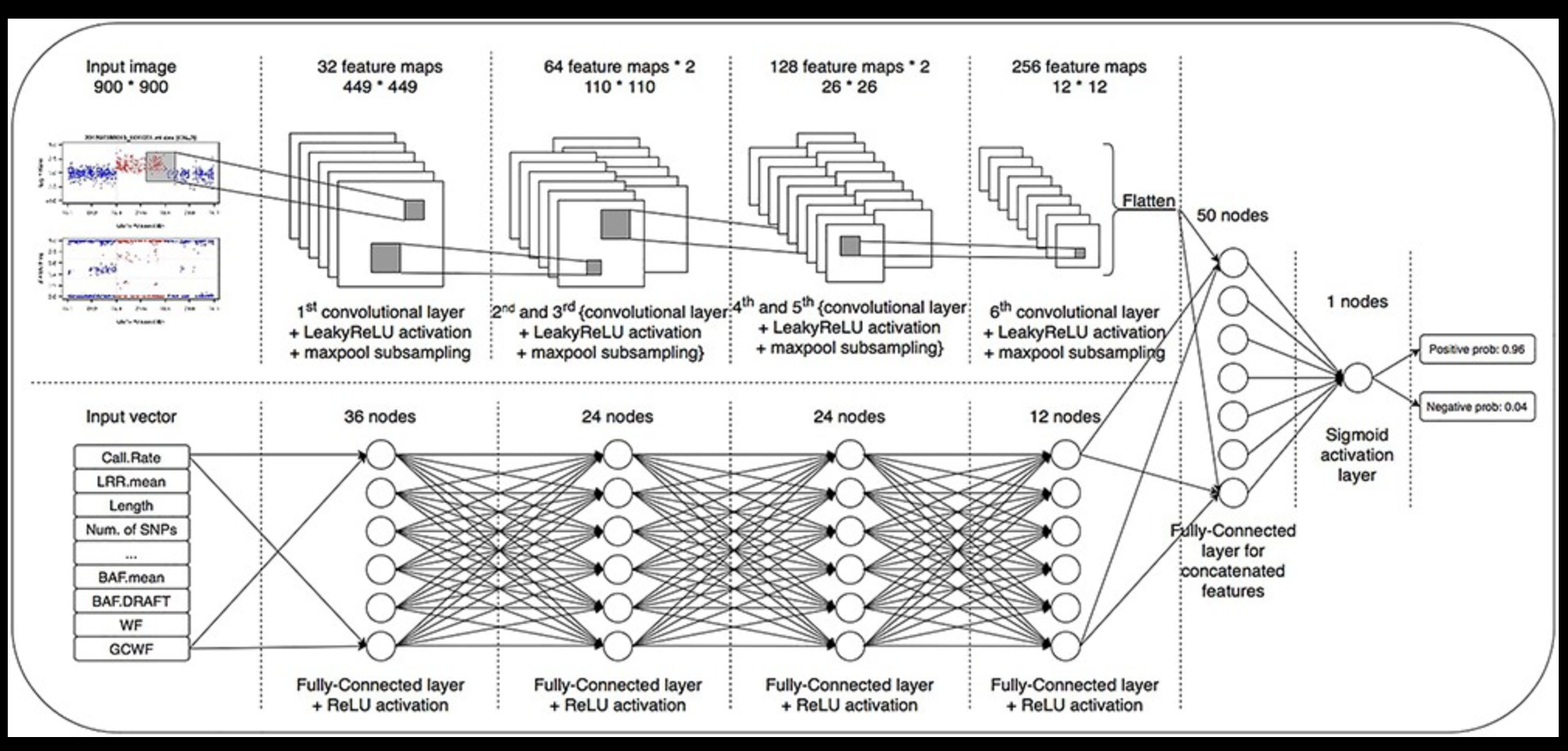
Brief. Bioinformatics |DeepCNV:一种用于验证拷贝数变异 (CNV) 的深度学习方法
题目:DeepCNV: a deep learning approach for authenticating copy number variations
杂志:Briefings in Bioinformatics
IF: 11.62
时间:05 September 2021
链接:https://doi.org/10.1093/bib/bbaa381
摘要
拷贝数变异(CNVs)是一类重要的变异,有助于许多疾病表型的发病机制。从基因组数据中检测CNVs仍然很困难,目前应用的大多数方法都存在不可接受的高假阳性率。一个常见的做法是在进一步的下游分析或实验验证之前,由人类专家手动审查原始的CNV调用以过滤假阳性。在这里,他们提出了DeepCNV,一个基于深度学习的工具,目的是在验证CNV调用时取代人类专家,重点是由最准确的CNV调用者之一PennCNV进行调用。深度神经网络算法的复杂性被超过10 000个专家评分的样本所丰富,这些样本被分成训练和测试集。变种的置信度,特别是对于CNVs,是阻碍CNVs与疾病联系起来的主要障碍。结果表明,DeepCNV增加了CNV调用的置信度,其最佳接收者操作特征曲线下面积为0.909,超过了其他机器学习方法。DeepCNV的优越性也通过实验性的湿实验室验证数据集进行了基准测试和确认。我们得出结论,DeepCNV获得的改进使假阳性结果和复制CNV关联结果的失败明显减少。
CNV图像产生的方法: PennCNV
为了直观地检查CNV调用并判断它们是否为真阳性,我们利用PennCNV提供的辅助可视化程序(visualize_cnv.pl)为CNV调用自动生成图像文件。对于每个CNV调用,它产生一个LRR散点图图像和一个BAF散点图图像。这些图涵盖了一个候选的CNV片段和它的周围区域。LRR图为该区域(候选CNV+其扩展区域)中的每个SNP基因分型画一个点,染色体位置为X坐标,LRR为Y坐标。BAF图以类似的图覆盖同一区域,但使用BAF作为Y坐标。对于这两张图,我们将候选CNV中的SNPs(点)涂成红色,将周围区域的SNPs涂成蓝色。然后,具有CNV调用专业知识的科学家根据视觉效果将它们标记为假阳性或真阳性。图1描述了样本图像。这些图像被用作DeepCNV的输入。图像中的像素范围从0到255。按照标准的图像缩放操作,我们通过将每个像素除以255来规范像素值,因此新的值在0和1之间,供DeepCNV使用。