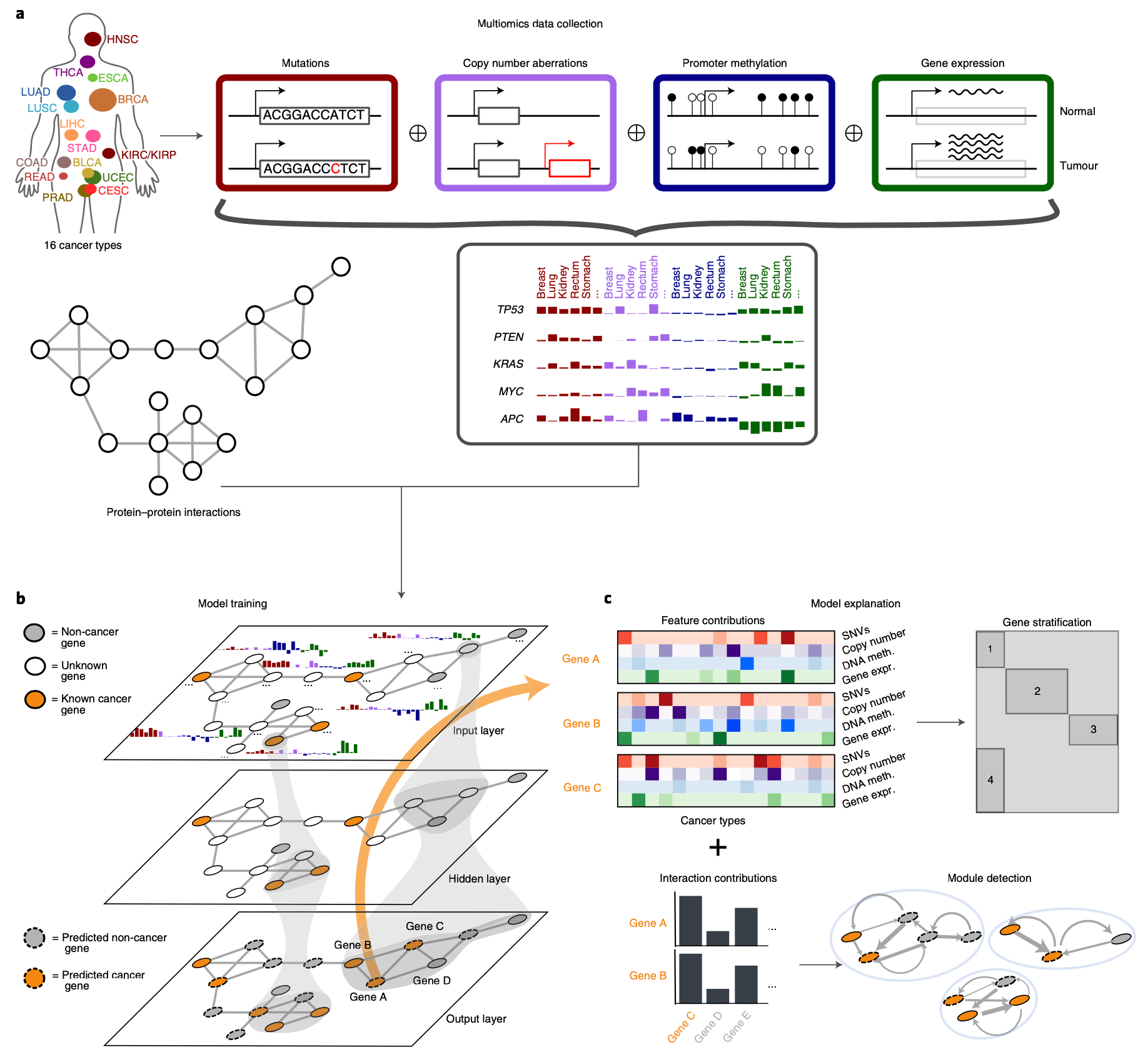
Brief. Bioinformatics | DeepHost:用卷积神经网络进行噬菌体宿主预测
题目:DeepHost: phage host prediction with convolutional neural network
杂志:Briefings in Bioinformatics
IF: 11.62
时间:22 September 2021
链接:https://doi.org/10.1093/bib/bbab385
摘要
新一代测序技术迅速扩大了已知的噬菌体基因组。与基于培养的方法不同,从下一代测序数据中发现的噬菌体的宿主仍未被描述。噬菌体基因组的高度多样性使得宿主分配任务具有挑战性。为了解决这个问题,我们提出了一个噬菌体宿主预测工具-DeepHost。为了将噬菌体基因组编码成矩阵,我们设计了一种基因组编码方法,应用各种间隔的K-mer对来容忍序列变化,包括插入、删除和突变。DeepHost应用卷积神经网络来预测宿主分类法。DeepHost在属级(72个分类群)的预测准确率为96.05%,在种级(118个分类群)的预测准确率为90.78%,比现有的噬菌体宿主预测工具高出10.16-30.48%,取得了与BLAST相当的结果。对于在BLAST中没有命中的基因组,DeepHost在属级获得了38.00%的准确率,在种级获得了26.47%的准确率,使其适用于与现有数据集同源序列较少的基因组。DeepHost是alignment-free的,它比BLAST快,特别是对大的数据集。DeepHost可在https://github.com/deepomicslab/DeepHost。
文章思路
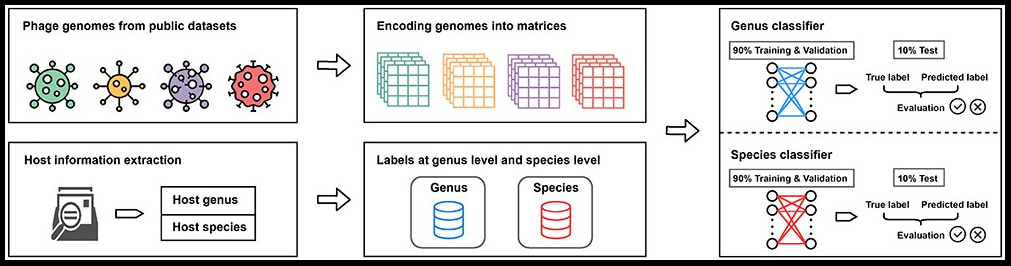
基因组序列编码方法:
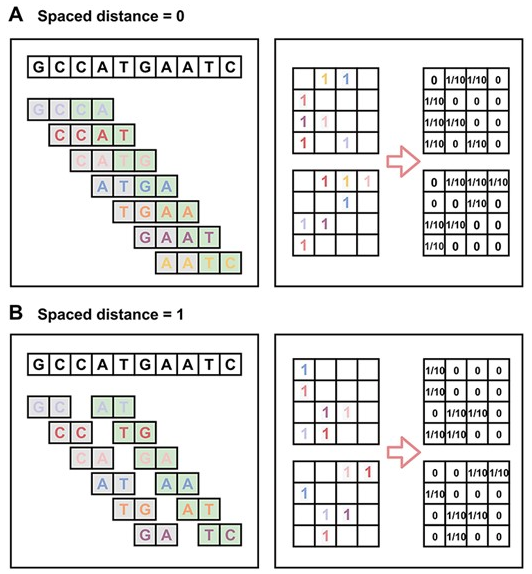
Illustration of the matrix construction process. Given a DNA sequence, all possible 2-mer pairs are collected with spaced distance of 0 (upper) and 1 (lower). For each distance, we construct two matrices.
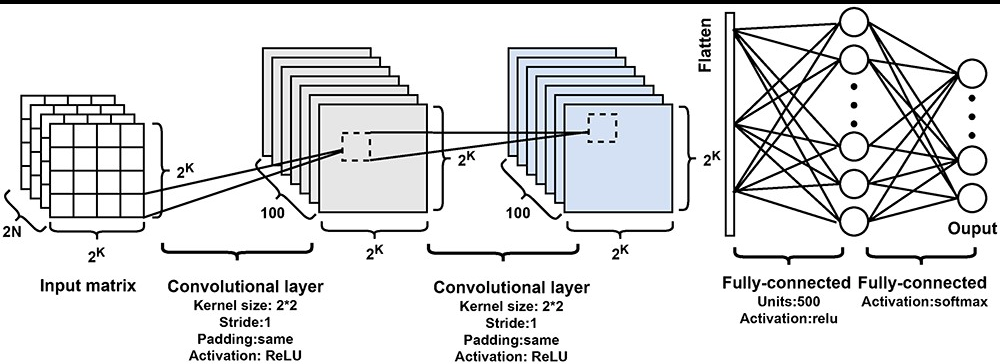
CNN 架构