单细胞技术是目前比较火热的一个领域,从单细胞转录组、基因组扩展到单细胞表观组,如scATAC-seq, scHiC, scmethylation等,促使着相关的分析工具也不断涌出。单细胞领域相关工具的开发大致有两个方向:一是从本质上如算法、统计学方法入手开发新的分析工具;一种是整合多种基础工具,开发出具有功能全面,易用等特征的工具。

MAESTRO(Model-based AnalysEs of Single-cell Transcriptome and RegulOme)是刘小乐实验室基于snakemake新开发的用于分析scRNA-seq和scATAC-seq的工具。MAESTRO主要是打包了一些工具和软件包,目前可以实现的功能包括:对scRNA-seq和scATAC-seq数据的比对、质量控制、细胞过滤、标准化、非监督聚类、差异分析、peak calling、细胞类型注释和转录调控分析。支持 Smart-seq2, 10x-genomics, Drop-seq, SPLiT-seq for scRNA-seq protocols; microfudics-based, 10x-genomics and sci-ATAC-seq for scATAC-seq protocols.
提供的有docker镜像,安装可以参考软件介绍:https://github.com/liulab-dfci/MAESTRO/
1 | wget https://repo.continuum.io/miniconda/Miniconda3-latest-Linux-x86_64.sh |
Installing the MAESTRO R package
1 | R |
另外依赖的cell ranger, RABIT/LISA等软件需要自行单独安装。
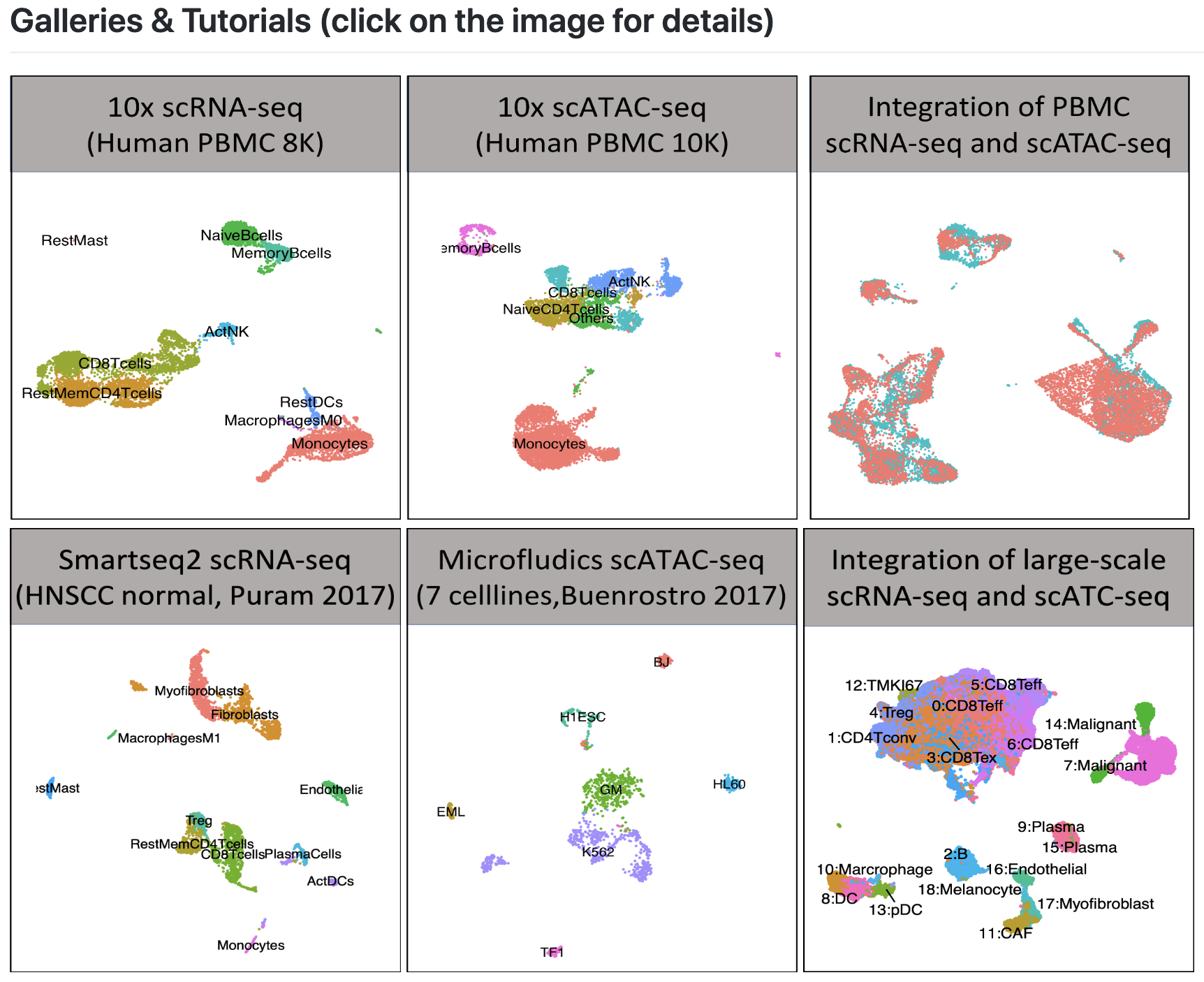
每种类型的数据的详细分析教程可以在原工具介绍网页点击下面每幅图查看
PS: 目前还没有看到相关文章发表