一句话评价
深度表征学习在疾病预测中的应用
文章信息
题目:DeepMicro: deep representation learning for disease prediction based on microbiome data
杂志:Scientific Reports
时间:7 April, 2020
链接: https://www.nature.com/articles/s41598-020-63159-5
figure
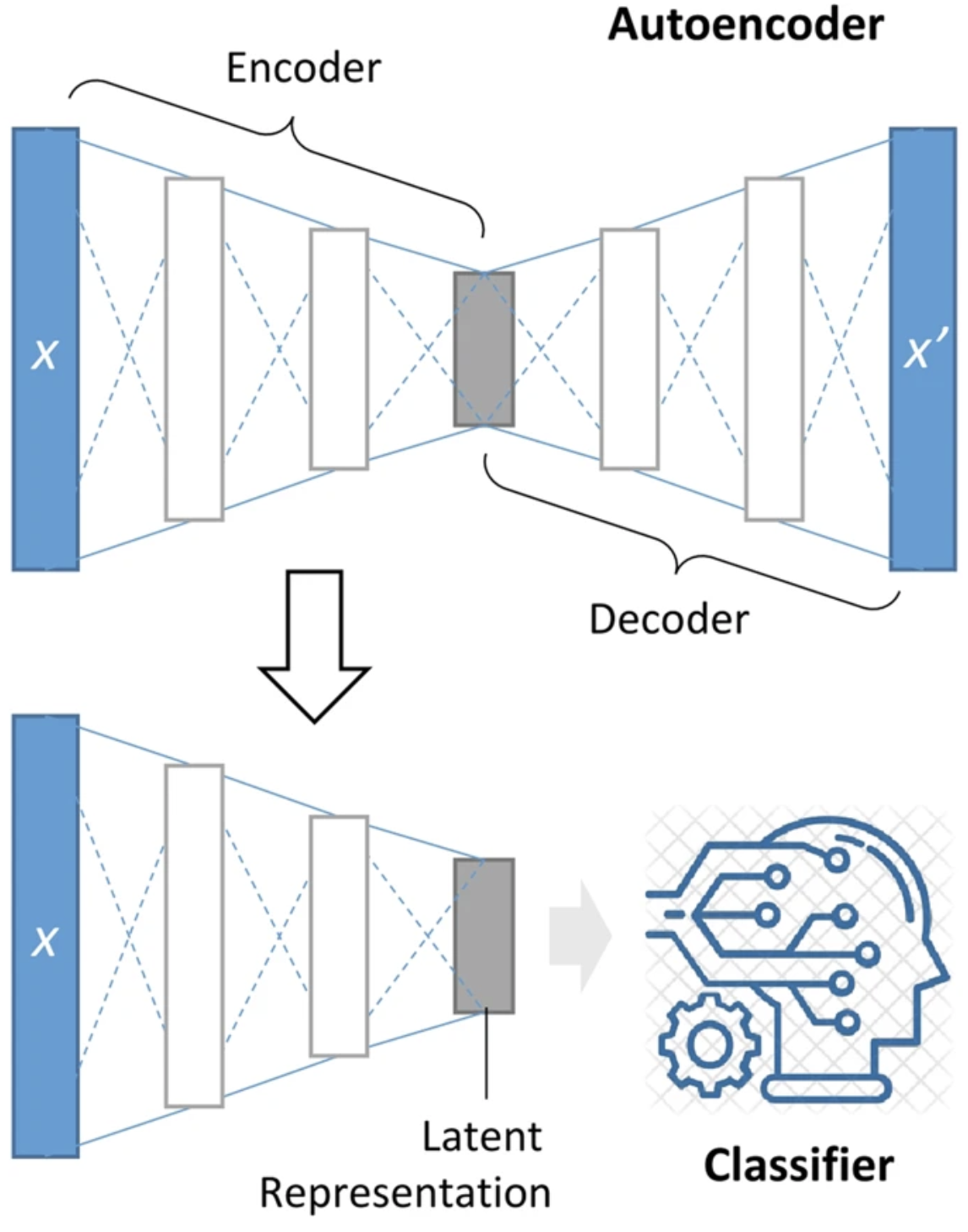
文章介绍:
微生物组学数据在人类健康和疾病领域中扮演着重要的作用,然而,微生物组学数据的高纬性,以及样本量低等特点使基于机器学习的预测算法面临极大的挑战。这篇文章基于深度表征学习框架,提出了DeepMicro的方法,可使用各种自动编码器成功的将高维数据转换为低维表示,加快了模型训练和超参数优化过程,在基本方法的基础上提高了8-30倍。在5个不同的数据集上的测试显示DeepMicro在疾病预测方面潜在应用。
DeepMicro的实现环境和使用的工具包有:
- Python 3.5.2
- Numpy 1.16.2,
- Pandas 0.24.2,
- Scipy 1.2.1,
- Scikt-learn 0.20.3,
- Keras 2.2.4
- Tensorflow 1.13.1
代码:https://github.com/minoh0201/DeepMicro
表征学习(representation learning)
机器学习算法的成功与否不仅仅取决于算法本身,也取决于数据的表示。数据的不同表示可能会导致有效信息的隐藏或是曝露,这也决定了算法是不是能直截了当地解决问题。表征学习的目的是对复杂的原始数据化繁为简,把原始数据的无效信息剔除,把有效信息更有效地进行提炼,形成特征,这也应和了机器学习的一大任务——可解释性。 也正是因为特征的有效提取,使得今后的机器学习任务简单并且精确许多。在我们接触机器学习、深度学习之初,我们就知道有一类任务也是提炼数据的,那就是特征工程。与表征学习不同的是,特征工程是人为地处理数据,也是我们常听的“洗数据”。 而表示学习是借助算法让机器自动地学习有用的数据和其特征。 不过这两个思路都在尝试解决机器学习的一个主要问题——如何更合理高效地将特征表示出来。