文章信息
题目:Application of machine learning techniques to tuberculosis drug resistance analysis
杂志:Bioinformatics, Data and text mining
IF: 5.610 (2019)
时间:21 November, 2018
链接:doi: 10.1093/bioinformatics/bty949
一句话评价
对比了不同的机器学习算法(LR,gradient tree boosting)在预测肺结核药物抗性中的性能
文章介绍
概述
- 及时识别结核分枝杆菌(MTB)对现有药物的耐药性对降低死亡率和防止现有抗生素耐药性的扩大至关重要。机器学习方法已被广泛应用于及时预测特定药物下MTB的耐药性和识别耐药标志物。然而,它们在耐药性预测和耐药性标志物识别方面还没有在全球多中心的MTB样本大队列上得到验证。
- 本文收集的样本来自6个大洲中的16个国家,共有13402个isolates,涉及到11种药物。
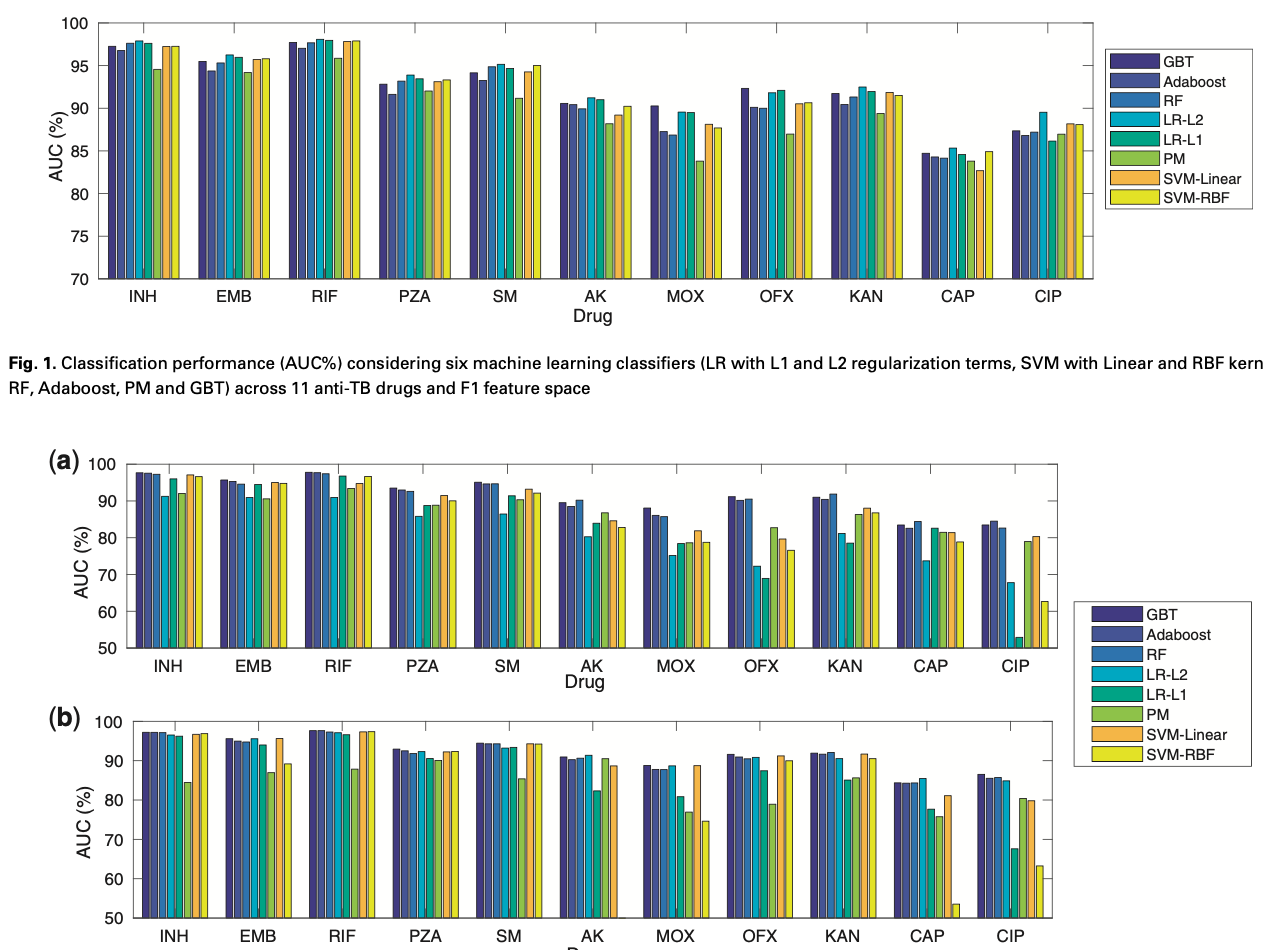
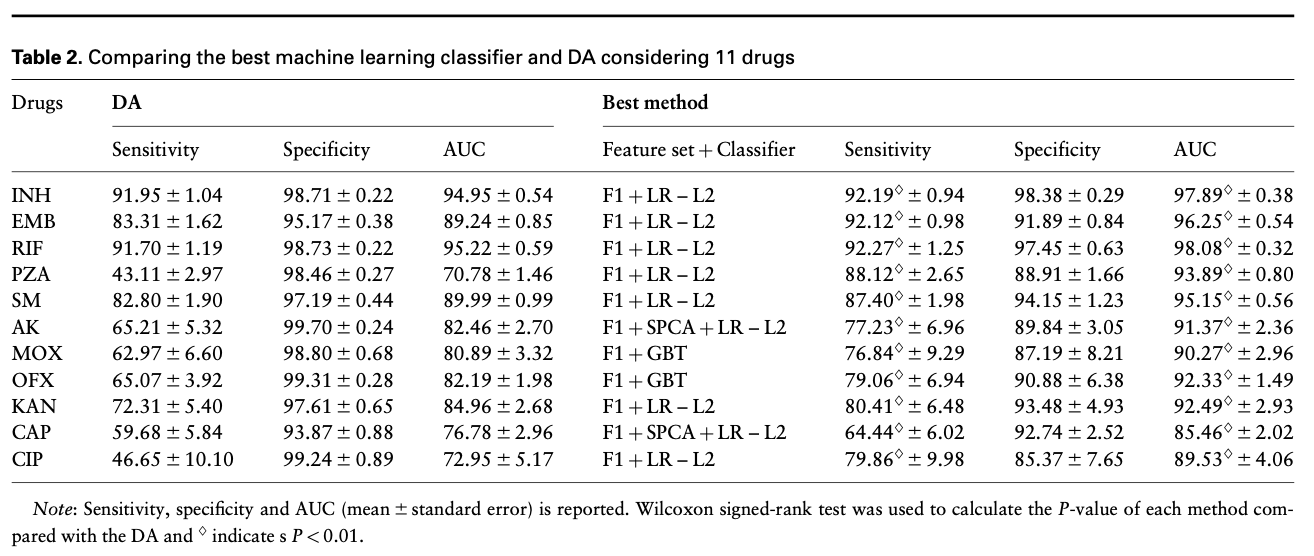
- 对比的机器学习方法有:SVM, LR, product-of-marginals (PM), gradient tree boosting, Adaboost, RF。其中LR和gradient tree boosting的性能比其他的算法好。
- 此外,根据突变位点的排序提供了一些潜在的研究靶标。并给出了文中所用的源码: http://www.robots.ox.ac.uk/davidc/code.php
方法
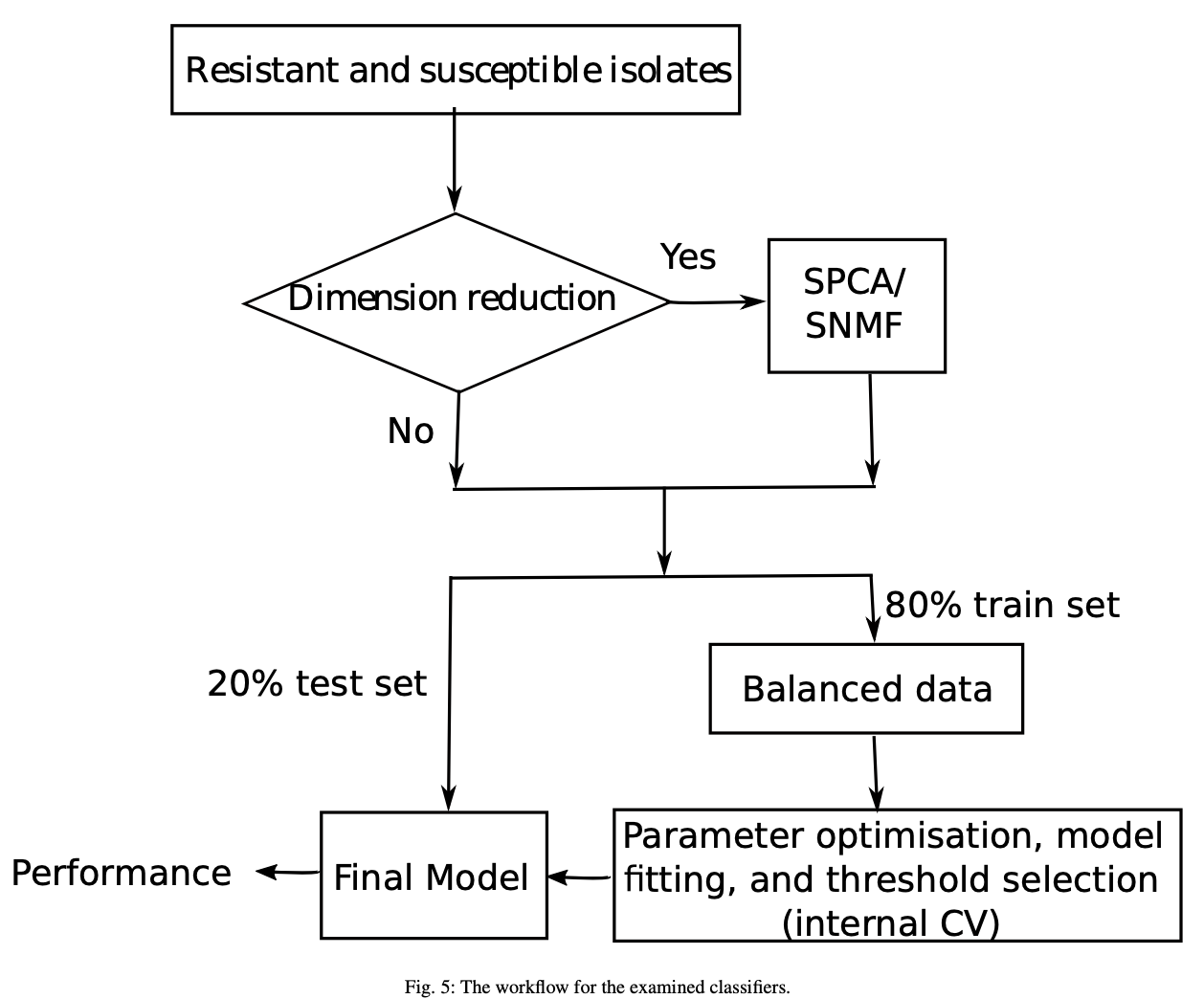
WGS数据分析
序列比对,call变异位点,过滤低质量的变异位点,最终保留5919个位点。
Baseline Methods
现有的基线方法是根据一些预先确定的变异库将药物分类为存在耐药性或不存在耐药性。直接关联(DA)的方法使用 “OR “规则来分类一个分离株对特定药物的抗药性:如果分离株的任何突变都是抗药性变异,则被标记为抗药性。否则,如果分离物中只有易感变异存在,则被归为易感。
变异库参考:Whole-genome sequencing for prediction of Mycobacterium tuberculosis drug susceptibility and resistance: a retrospective cohort study. Lancet Infect. Dis
线性降维
使用PCA对特征(变异位点)进行降维
分类器方法
SVM, LR, product-of-marginals (PM), gradient tree boosting, Adaboost, RF
结果和结论
数据描述
- 总分离株(isolates)的数量:13402
- 变异位点:是与之前鉴定到的23个抗性基因相关的位点,共有5919个位点
- 11个药物的标签信息(Resistant/Susceptible), 标签信息不平衡,所有11种药物的易感性分离株均大大高于耐药性分离株。
特征空间(Feature Spaces)
为了评估不同分类器的性能,文中考虑了三个特征集。1) F1是基线特征空间,即在23个候选基因内发现的所有变异。2) F2是根据另一篇文章(https://pubmed.ncbi.nlm.nih.gov/26116186/)中列出的预定抗药性相关变异。3) F3是F1的子集,仅包括特定药物的抗药性相关基因(每种药物特有的抗药性决定因素基因,也是根据之前的一篇文章(https://pubmed.ncbi.nlm.nih.gov/26116186/)。
训练和测试
分类模型是通过训练一个平衡的训练数据集,然后在不平衡的数据集上进行测试来执行的。并运行超过100次的5-kfold的交叉验证。在每个fold, 选取20%数据作为测试集,剩余的80%的训练集,将易感样本随机分选,使耐药和易感样本的数量相等,然后拆分训练集和验证集(80%:20%)。
模型评估参数: accuracy, sensitivity, spe- cificity, F1-score 和ROC curve的 area under curve (AUC)
分类结果
突变排序
选取性能最好的模型,然后提取前10个对每个药物有特有抗性的features。
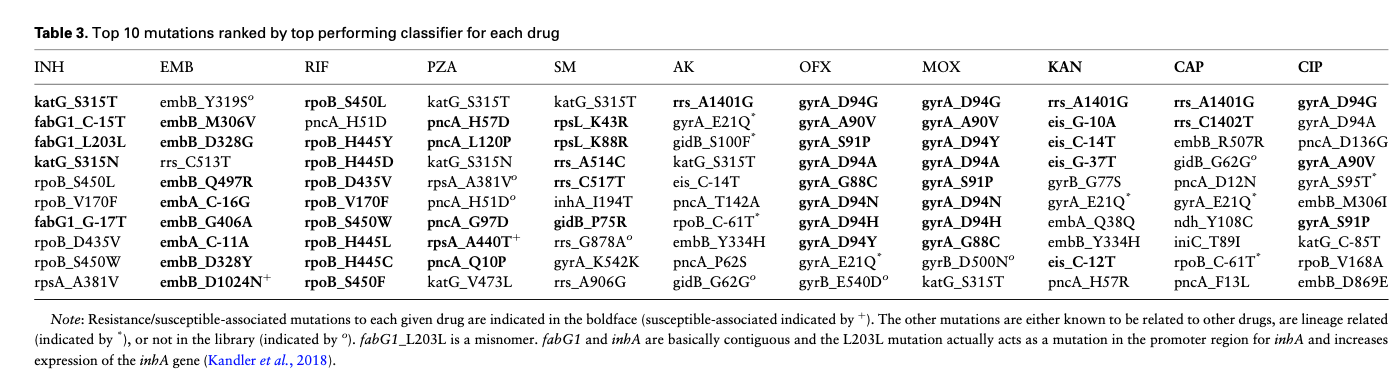
创新性和意义
- 样本量大,最后提供了药物相关的一些潜在靶标
- 文章思路:首先是WGS变异分析,然后选取之前研究的25个抗性基因相关的变异位点,再通过降维方法进一步减小特征的维度,最后比较不同的模型对不同组合提取的特征数据集的性能,并通过特征选择给出了每种药物特有的潜在变异位点。