题目:Multi-omic machine learning predictor of breast cancer therapy response
杂志:Nature
IF: 49.96
时间:7 Dec. 2021
链接:https://www.nature.com/articles/s41586-021-04278-5
代码:
- https://github.com/micrisor/NAT-ML
- https://github.com/cclab-brca/neoadjuvant-therapy-response-predictor
一句话概括
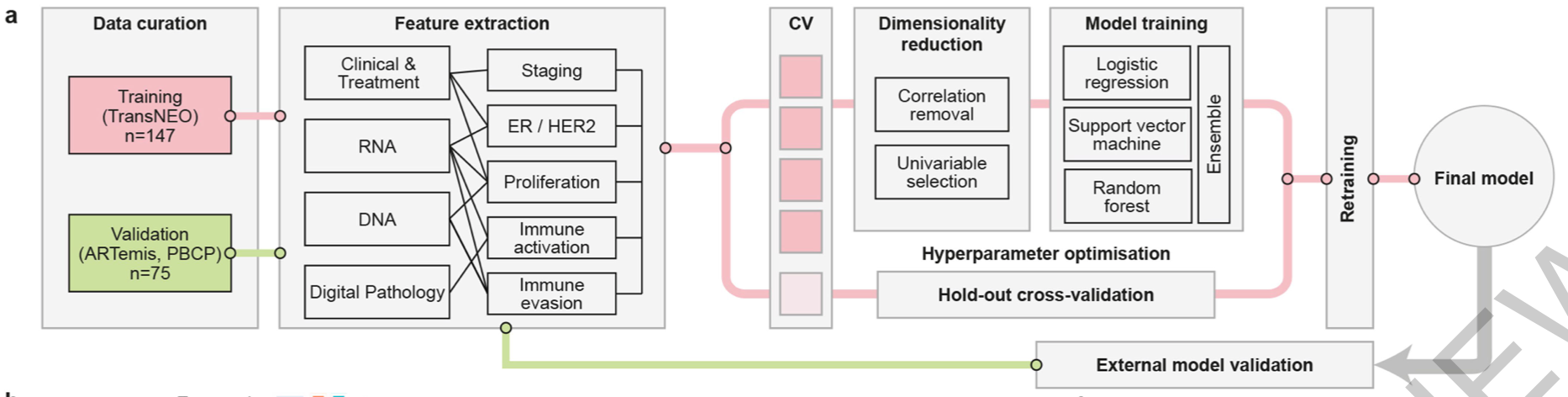
使用逻辑回归,SVM和随机森林的组合机器学习方法,结合临床数据、数字病理学数据、基因组和转录组学数据,对乳腺癌治疗的反应进行预测
###摘要
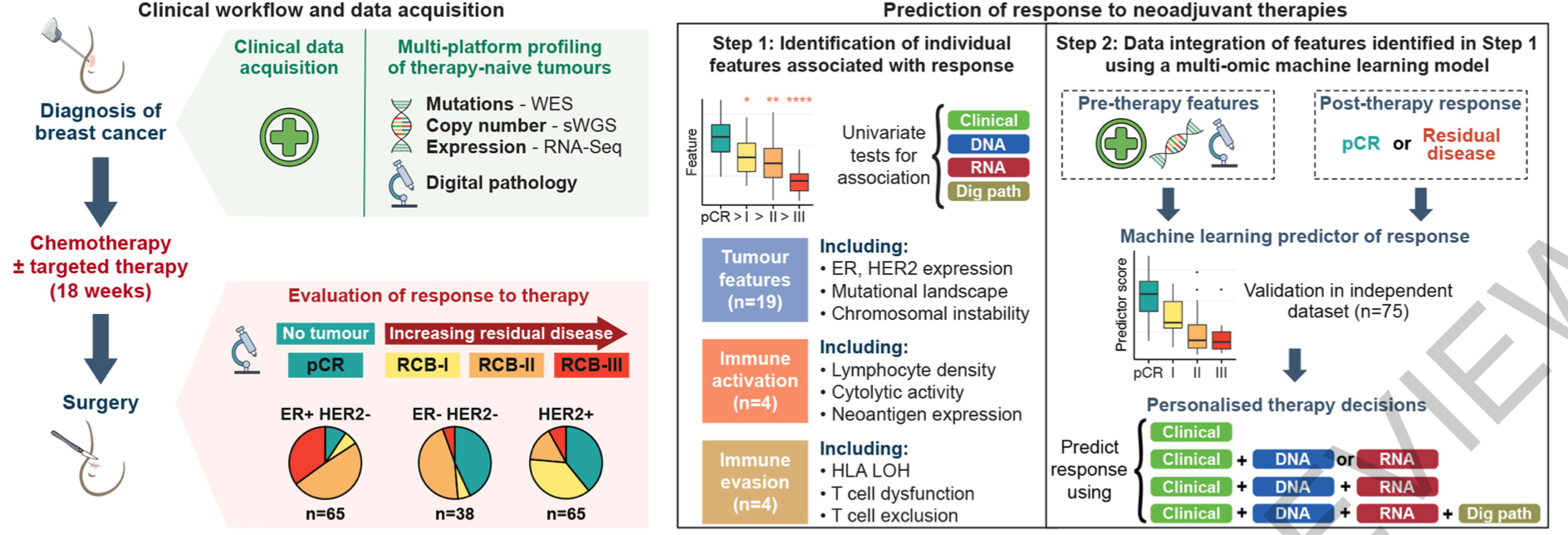
乳腺癌是由恶性细胞和肿瘤微环境组成的复杂生态系统。这些肿瘤生态系统的组成和其中的相互作用有助于细胞毒性治疗反应。构建细胞毒性治疗反应的预测器还有待研究。这篇文章收集了168名患者在手术前接受化疗+/-HER2靶向治疗的乳腺肿瘤治疗前活检的临床、数字病理学、基因组和转录组资料。然后将手术时的病理终点(完全反应或残余疾病)与这些诊断性活检的多组学特征相关联。他们发现,对治疗的反应是由预处理的肿瘤生态系统调节的,其多组学景观可以用机器学习整合到预测模型中。治疗后残余疾病的程度与治疗前的特征单调相关,包括肿瘤突变和拷贝数变异、肿瘤增殖、免疫浸润和T细胞功能紊乱和排斥。将这些特征结合到一个多组学机器学习模型中,可预测外部验证队列(75名患者)的病理完全反应,AUC为0.87。总之,对治疗的反应是由通过数据整合和机器学习捕获的整个肿瘤生态系统的基线特征决定的。这种方法可用于开发其他癌症的预测器。