单细胞发展至今,多组学整合是其发展的一个方向,关于scRNAseq与scATACseq整合的方法也有很多文章报道,这篇文章是由表观学大牛任兵实验室开发的,主要优点体现在通量上,可用于并行分析数百万个单细胞转录组和可访问染色质。
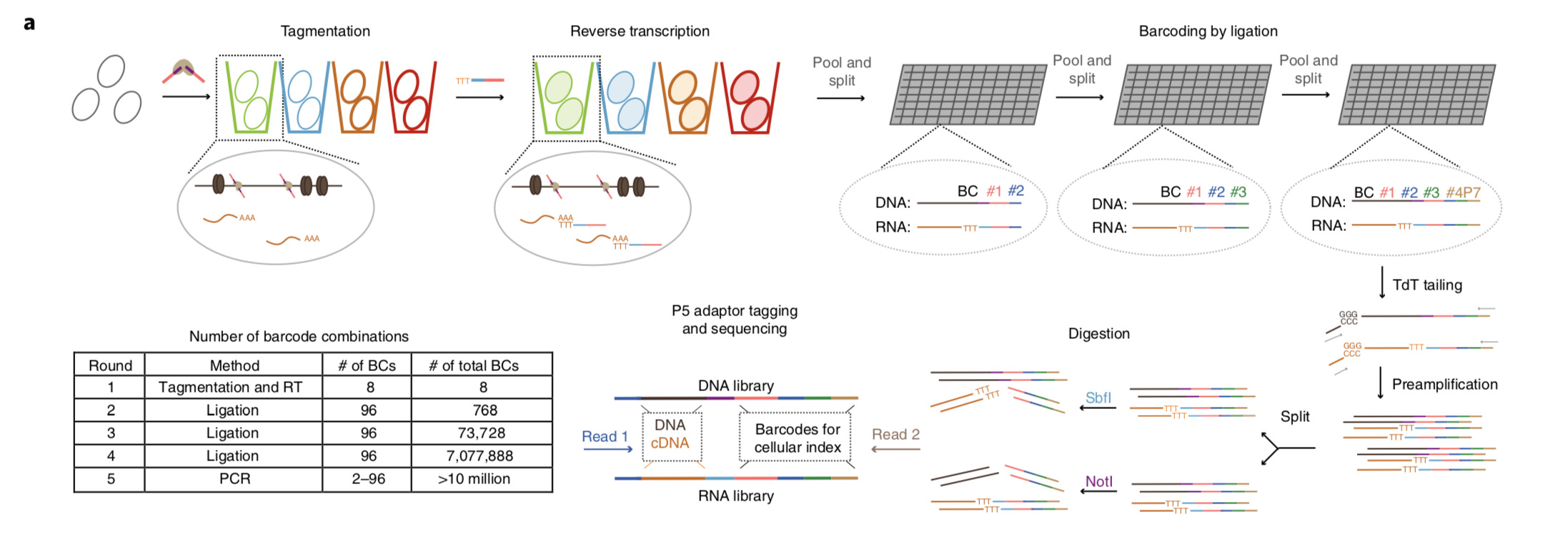
单细胞发展至今,多组学整合是其发展的一个方向,关于scRNAseq与scATACseq整合的方法也有很多文章报道,这篇文章是由表观学大牛任兵实验室开发的,主要优点体现在通量上,可用于并行分析数百万个单细胞转录组和可访问染色质。

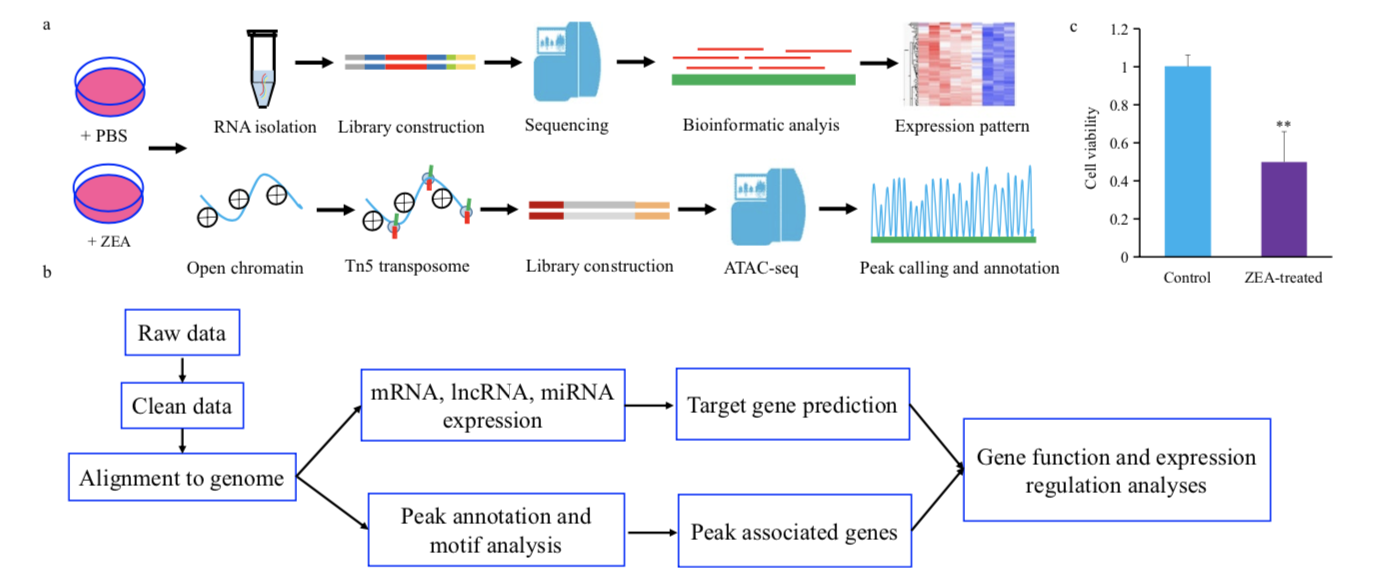
这篇文章主要想研究非编码RNA与染色质可及性对玉米赤霉烯酮对猪小肠上皮细胞的影响,思路较简单,分析也过于简单,文章也没有得到特定的结论。思路如下图b所示,分析ATAC-seq得到peaks,然后注释peaks的基因,分析RNA-seq得到miRNA,lncRNA,然后预测这些非编码RNA的靶基因,并将非编码RNA的靶基因与peak注释到的基因整合,进行功能注释。
一句话评价
深度学习在宏基因组分析的物种鉴定中的应用
文章信息
题目:DeepMicrobes: taxonomic classification for metagenomics with deep learning
杂志:BioRxiv
时间:Jul,8, 2019
之前分享过哈尔滨医科大学李霞老师实验室开发的lncRNA-chromatin相互作用数据库——LnChrom (http://biocc.hrbmu.edu.cn/LnChrom/index.jsp) 。这次详细解读数据库背后所做的分析,同时总结文章的思路、结论和成果以及对其他研究的启发。

常见的可视化基因组track的Bioconductor package有rtracklayer 和 Gviz。rtracklayer提供了交互的基因组浏览器和相关tracks的注释;Gviz可以用于tracks覆盖度和注释的可视化。trackViewer是一个更轻量级的工具,且可以产生交互的可视化结果。可用于RNA-seq,ChIP-seq/ATAC-seq,miRNA-seq,DNA-seq,methylation等各种NGS组学数据的覆盖度和注释tracks的可视化展示,而且可以绘制发表级的棒棒糖图和蒲公英图。
先展示下极好看可视化效果
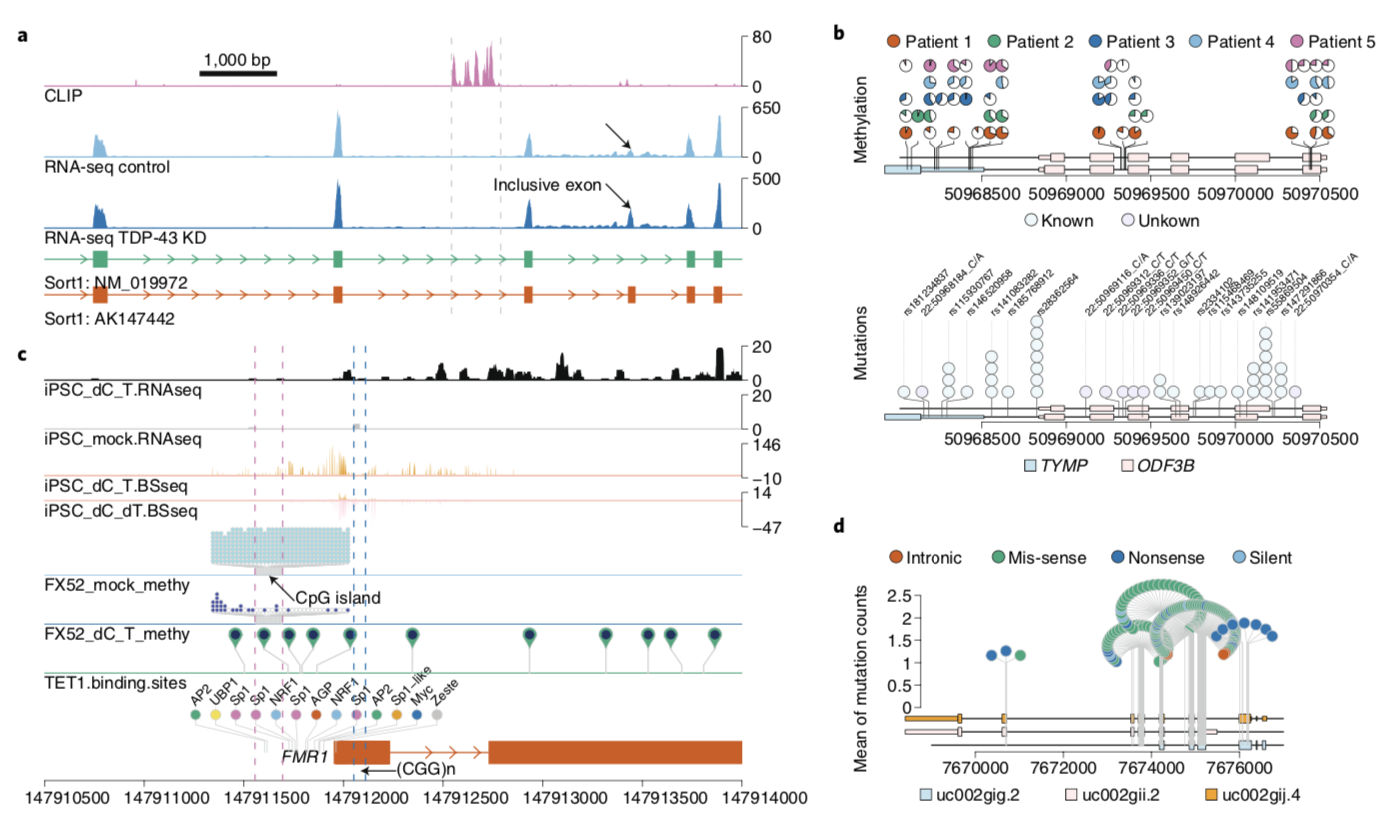
使用方法可以参考文档: