
方法概述:
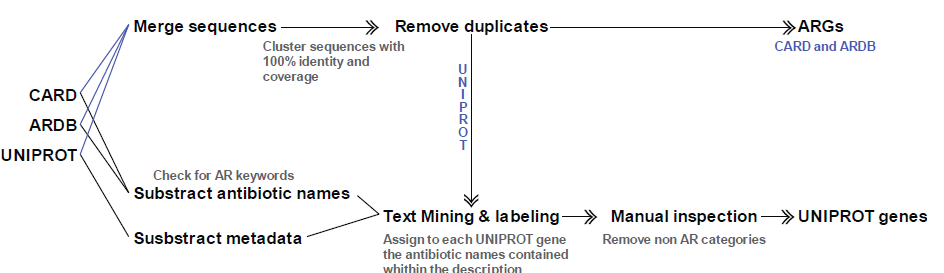
从CARD,ARDB和UNIPROT 3个数据库分别提取抗性基因(antibiotic resistance genes,ARGs), 3个数据库分别提取到2161,2290和28108个抗性基因。然后对3个数据库的抗性基因进行注释和分类,CARD和ARDB共鉴定到102个抗生素,包括30个抗生素分类。UNIPROT通过文本挖掘注释,然年再结合CARD和ARDB两个数据库验证。
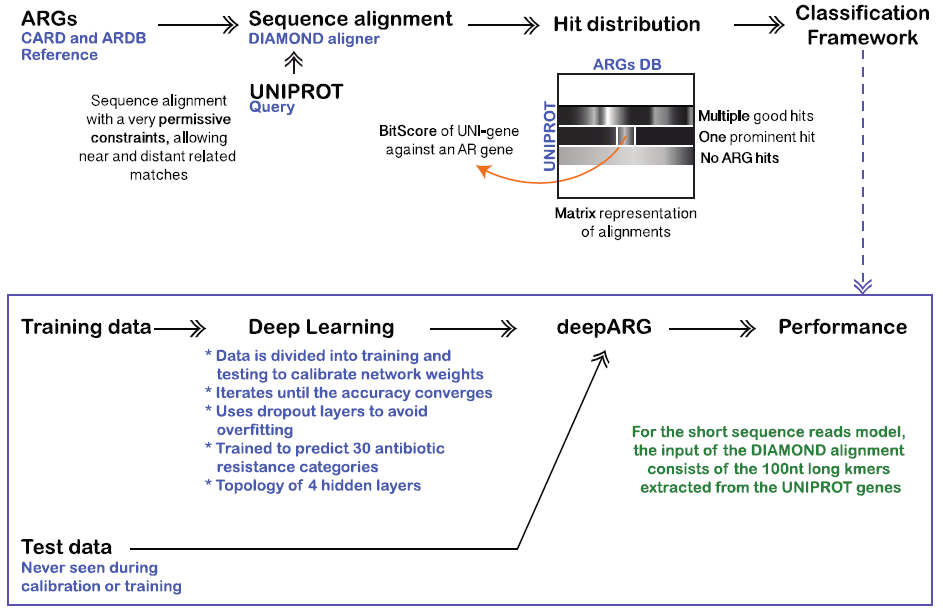
得到经过前处理后的数据,利用深度神经网络训练模型,凭借Python中的Theano库的Lasagne模块实现模型训练。分别对短读长序列和长基因序列分开训练,得到deepARG-SS 和 deepARG-LS。
评估模型通过两个方法:一是UNIPROT的70%数据作为训练数据,30%作为测试验证数据;二是借助MEGARes数据库的数据作为独立的验证数据集,评估模型的表现。
数据来源:
数据来自3个数据库:CARD,ARDB和UNIPROT
数据前处理
模型训练
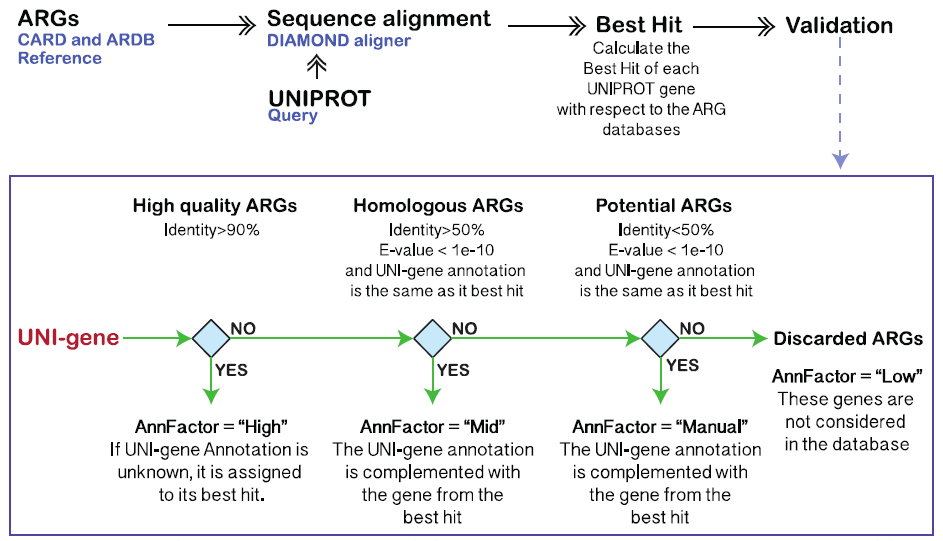
模型验证
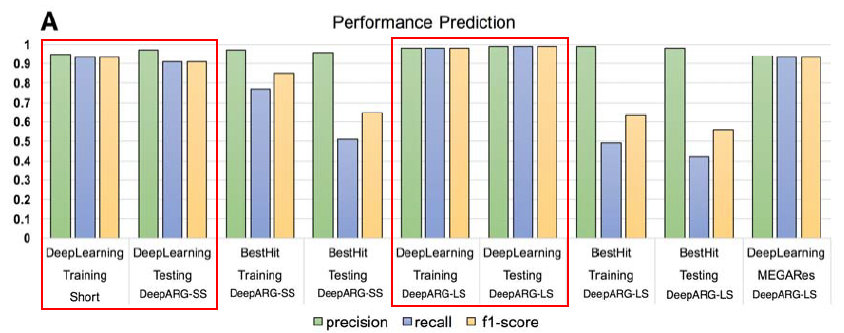
从模型验证的结果看,似乎模型的效果太好了,不知道是否有过拟合的现象。
实现代码
github代码打不开:
https://gaarangoa@bitbucket.org/gaarangoa/deeparg-ss.git
网站在线分析: